中国对虾“黄海1号”具有生长快、抗逆性强的优良特性。这一新品种的选育成功,为中国对虾养殖业的可持续发展和处于困境中的中国对虾养殖的“二次创业”提供了重要的品种保障。中国对虾的人工选育工作已取得了一系列研究进展,包括利用RAPD、AFLP和微卫星技术分别对不同地理群体和选育群体不同家系进行遗传多样性分析(孟宪红等,2004;何玉英等,2004;张天时等,2005;Liu et al,2006;岳志芹等,2005);为进行数量性状位点(QTL)定位和标记辅助选择,采用RAPD、SSR和AFLP技术进行了中国对虾遗传图谱的构建(孙昭宁等,2006;王伟继等,2006;岳志芹等,2004;Li et al,2006;李健等,2008);利用分子生物学的方法获得与中国对虾生长性状相关的候选标记(刘萍等,2007;何玉英等,2007)。这些研究结果为中国对虾标记辅助育种提供了理论依据,大大加快了中国对虾的标记辅助育种的进程。而关于决定人工选育中国对虾“黄海1号”优良经济性状的分子机制的研究尚未开展。为了深入探讨中国对虾“黄海1号”的优良遗传特性,李吉涛等(2009)构建了中国对虾“黄海1号”的血细胞和肌肉组织cDNA文库,为进一步筛选血液和肌肉特异表达基因奠定分子基础。
本实验所用中国对虾取自中国对虾“黄海1号”下营良种推广基地,活体采取血液和肌肉组织,立即置于液氮中,以备提取RNA。
从电泳结果可以看出(图4),18S、28S rRNA条带清晰,总RNA较完整。分光光度计检测,血细胞总RNA样品的OD260/OD280为1.82,肌肉总RNA样品的OD260/OD280 为1.85,说明总RNA纯度较高。纯化mRNA是获得高质量的cDNA文库的关键,分离纯化的mRNA琼脂糖电泳检测(图5),可见呈smear现象,纯度和大小符合理论值,可以用于构建高质量cDNA文库。

图4 中国对虾血细胞和肌肉总RNA电泳图
1:从血细胞提取的总RNA;2:从肌肉组织提取的总RNA
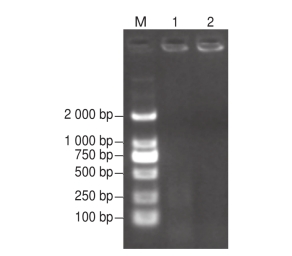
图5 中国对虾血细胞和肌肉mRNA电泳图
M: DL2000相对分子质量标准;1:血细胞mRNA;2:肌肉mRNA
采用SMART技术,用50 ng血细胞或肌肉mRNA反转录成单链cDNA,通过LD-PCR扩增获得双链cDNA,取5mL PCR产物在1.0%琼脂糖凝胶电泳分析,相对分子质量大小分布于0.1~10 kb,大小分布均匀(图6),满足cDNA文库的构建要求。cDNA片段用CHROMA SPINTM-400分离柱分级分离,取3mL分离产物进行电泳,发现cDNA片段从第5管后出现,收集第5~8管可以确保cDNA片段大于400 bp,可以用于构建cDNA文库。
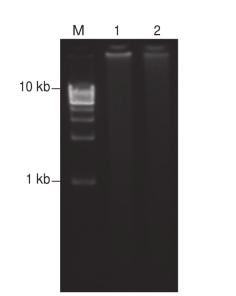
图6 中国对虾血细胞和肌肉双链cDNA电泳图
M:13103相对分子质量标准;1:血细胞双链cDNA;2:肌肉双链cDNA
收集的cDNA片段与λTriplEx2载体重组,体外包装和文库贮存按Promega公司的Packagene®Lambda DNA包装系统说明书进行。我们发现cDNA和载体的摩尔比在1:1时能产生有效的重组体。包装后血细胞cDNA文库容量为2.36×106,肌肉文库容量为0.77.3 106,文库扩增后,血细胞文库滴度为5.6.3 109 pfu/mL,肌肉文库滴度为3.0.3 109 pfu/mL,经IPTG诱导检测,重组率均达到98%以上;从各文库随机取出10个清晰的噬菌斑进行PCR扩增,琼脂糖凝胶电泳检测,其插入片段长度为400~2.0.0 bp(图7和图8)。证明两文库质量较好。

图7 中国对虾血细胞cDNA文库噬菌斑的PCR扩增产物电泳图
M: DL2000相对分子质量标准;1-10:代表10个克隆的PCR产物

图8 中国对虾肌肉cDNA文库噬菌斑的PCR扩增产物电泳图
M: DL2000相对分子质量标准;1-10:代表10个克隆的PCR产物
构建cDNA文库并从中筛选目的基因是一种十分有效的寻找新基因的手段,因为cDNA文库是众多cDNA序列的集合,不包含繁复的基因组序列,直接编码氨基酸,因此包含了大量基因资源和信息,这使得基因的寻找更容易、快捷,并且使功能克隆新基因更有效。评价一个cDNA文库主要考虑三个因素:① 要有足够的克隆数,能够包含目的序列,特别是那些来自低丰度mRNA;② cDNA小片段(,500 bp)插入的克隆数较少;③ 由近于全长的mRNA拷贝cDNA插入组成,以便能获得全长基因。SMART为获得全长cDNA提供了技术保障。本研究采用SIMART技术成功构建的中国对虾“黄海1号”血细胞和肌肉cDNA文库,原始文库容量分别为2.36.3 106.0.77 3106,重组率达到98%以上,扩增后文库的滴度分别达5.6.109 pfu/mL、3.0.109 pfu/mL,大大超过了cDNA文库构建的基本要求。cDNA文库的插入片段长度分布于400~2.0.0 bp,说明两文库的质量较高,为进一步筛选、克隆新基因提供了重要资源。目前,作者已成功从肌肉文库中克隆了肌钙蛋白I基因cDNA全长,已被基因文库收录(GenBank登录号:FJ609301)。
随着对虾育种工作的深入,与对虾经济性状相关的重要基因的筛选、克隆与功能分析已经成为研究热点之一。cDNA文库是当前发现新基因和研究基因功能的基本工具,因此构建对虾特定部位组织的cDNA文库,可以从中筛选和克隆与对虾各项生理活动相关的功能基因,为进一步进行基因功能验证以及应用奠定基础。关于中国对虾cDNA文库的构建工作已有一些报道,李太武等(1998)用成虾头胸部(去头胸甲及胸部附肢)作为实验材料构建了cDNA文库,获得了一定数量的克隆。张晓军等(2005)构建了中国对虾6种组织(血液、眼柄、卵巢、雌虾头胸部、雄虾头胸部和三倍体对虾头胸部组织)的cDNA文库。王维新等(2004)构建了中国对虾鳃细胞全长cDNA文库。本研究以人工选育的海水养殖动物新品种中国对虾“黄海1号”为材料,构建了血细胞和肌肉的全长cDNA文库,为有效保存种质资源和后续功能基因的开发奠定了重要基础。
[1] 邓景耀 .海洋渔业生物学[M].北京:农业出版社,1991.
[2] 邓景耀,叶昌臣,刘永昌.黄渤海的中国对虾及其资源管理[M].北京:海洋出版社,1990.
[3] 邓景耀,朱金声 .渤海湾对虾产卵场调查[J] .海洋水产研究,1983,5:17-23.
[4] 邓景耀,庄志猛 .渤海对虾补充量变动的分析及对策研究[J].中国水产科学,20.1.7(4):125-128.
[5] 邓景耀 .对虾放流增殖研究[J].海洋渔业,1997,1:1-6.
[6] 杜玉珍,高锋 . 用cDNA-AFLP技术筛选新生牛软骨无血管区的高表达基因[J].中国生物化学与分子生物学报,20.8.24(1):55-59.
[7] 何玉英,刘萍,李健,等 .中国对虾人工选育群体第一代和第六代遗传结构分析[J].中国水产科学,20.4.11(6):572-575.
[8] 何玉英,刘萍,李健,等.中国对虾与生长性状相关SCAR标记的筛选[J].海洋与湖沼,20.7.38(1):42-48.
[9] 季士治,王伟继,雷霁霖,等 .大菱鲆AFLP分析体系的建立[J].海洋水产研究,20.7.28(1):6-12.
[10] 李朝霞 .中国对虾人工选育群体遗传结构分析及遗传连锁图谱的构建[D] .中国海洋大学博士论文,2006.
[11] 李法君,傅洪拓,王亮晖,等.日本沼虾AFLP反应体系的建立[J].生物技术,20.8.18(1):36-39.
[12] 李吉涛,李健,陈萍,等 .中国明对虾“黄海1号”血细胞和肌肉cDNA文库的构建[J].中国水产科学,2009,16:781-785.
[13] 李健,陈萍,刘萍,等 . cDNA-AFLP分析方法在中国对虾中的应用[J].中国海洋大学学报:自然科学版,2009,1208-1212.
[14] 李健,刘萍,何玉英,等 . 中国对虾快速生长新品种“黄海1号”的人工选育[J].水产学报,20.5.29(1):1-5.
[15] 李健,刘萍,王清印,等 .中国对虾遗传连锁图谱的构建[J].水产学报,2008,32(2):161-173.
[16] 李思发 .鱼类选育群体遗传性能的保护[J] .水产学报,19.8.2(3):283.2.0 .
[17] 李思发 .主要养殖鱼类种质资源研究进展[J] .水产学报,19.3.17(4):344-358.
[18] 李太武,相建海,刘瑞玉.中国对虾cDNA文库的构建[J] .动物学报,1998,44(2):237-238.
[19] 刘萍 .DNA标记技术在海洋生物种质资源开发和保护中的应用[J] .中国水产科学,2000,7:86-89.
[20] 刘萍,何玉英,孙昭宁,等 .中国对虾生长性状相关遗传标记的筛选与克隆[J] .海洋水产研究,20.7.28(2):1-6.
[21] 刘萍,孔杰,石拓,等 .中国对虾黄渤海沿岸群亲本及子一代RAPD分析[J],海洋水产研究,20.0.21(1):13-21.
[22] 刘萍,孔杰,石拓,等 .中国对虾黄渤海沿岸地理群的RAPD分析[J] .海洋学报,20.0.22(5):87-94 .
[23] 刘萍,李健,何玉英,等 .中国明对虾种质资源研究现状与保护策略[J] .海洋水产研究,20.4.25(5):80-85.
[24] 刘萍,麦明,孔杰,等 .中国对虾染色体制备及染色体形态的研究[J] .海洋科学,1994,1.33.36 .
[25] 刘萍,麦明,王清印,等 .中国对虾染色体及核型分析[J] .海洋水产研究,1992,13:29-34.
[26] 刘萍,孟宪红,何玉英,等 .中国对虾黄、渤海3个野生地理群遗传多样性的微卫星DNA分析[J] .海洋与湖沼,2004,35:252-257.
[27] 刘萍,孟宪红,孔杰,等 .中国对虾微卫星DNA多态性分析[J] .自然科学进展,20.4.14(2):150-155.
[28] 刘萍 .中国对虾染色体研究近况[J] .海洋水产研究,1987,8:88.
[29] 刘瑞玉 .黄海及东海经济虾类区系的特点[J] .海洋与湖沼,19.9.2(1):35-42 .
[30] 刘瑞玉 .中国大百科全书:农业卷(Ⅰ)[M] .北京:中国大百科全书出版社,1991. [31] 刘振辉,孔杰,孟宪红,等 .中国对虾两个不同地理群遗传结构的RAPD分析[J] .应用与环境生物学报,20.0.6(5):440-443.
[32] 孟宪红,马春燕,刘萍,等 .黄渤海中国对虾6个地理群的遗传结构及其遗传分化[J] .高技术通讯,2004,4:97-102.
[33] 石拓,孔杰,刘萍,等 .中国对虾遗传多样性的RAPD分析—朝鲜半岛西海岸群体的DNA多态性[J] .海洋与湖沼,19.9.30(6):609 -615 .
[34] 石拓,孔杰,庄志猛,等 .中国对虾遗传多样性的RAPD分析[J] .自然科学进展,20.1.11(4):360.3.4 .
[35] 石拓,孔杰,庄志猛,等 .中国对虾遗传多样性分析—朝鲜半岛西海岸群体的DNA多态性[J] .海洋与湖沼,19.9.30(6):609-615.
[36] 石拓,庄志猛,孔杰等 .中国对虾遗传多样性的RAPD分析[J] .自然科学进展,20.1.11(5):360-364.
[37] 孙昭宁,刘萍,李健,等 .RAPD和SSR两种标记构建的中国对虾遗传连锁图谱[J] .动物学研究,20.6.27(3):317-324.
[38] 王金星,赵小凡 .对虾组织蛋白质和同工酶表型及其在病虾中的变化[J] .海洋科学,1995,3:46-51.
[39] 王清印,李健,孔杰。养殖对虾的遗传改良“世界水产养殖科技大趋势”[M] .北京:海洋出版社,2003.
[40] 王维新,史成银,黄倢 .中国对虾鳃细胞全长cDNA文库的构建[J] .20.4.25(5):6-11.
[41] 王伟继,孔杰,董世瑞,等 .中国对虾AFLP分子标记遗传连锁图谱的构建[J] .动物学报,20.6.52(3):575-584.
[42] 王伟继,孔杰 . ISSR-PCR技术在对虾中应用初步研究[J] .海洋水产研究,2002,23(1):1-4.
[43] 相建海,刘旭东 .中国对虾种群生化遗传学研究 .见:曾呈奎,相建海主编 .海洋生物技术[M] .济南:山东科学技术出版社,1998,269-282.
[44] 相建海 .中国对虾染色体研究[J] .海洋与湖沼,1988,19(3):205-209.
[45] 徐鷉,周岭华,相建海 .中国对虾微卫星DNA的筛选[J] .海洋与湖沼,2001,32(3):255-259.
[46] 徐鷉,周令华,田丽萍,等 .从中国对虾ESTs中筛选微卫星标记的研究[J] .水产学报,20.3.27(3):213-218.
[47] 许再复 .生物多样性保护的现状趋势与展望:未来十年的生物科学[M] .上海:上海科学技术出版社,1991,88-100.
[48] 岳志芹,王伟继,孔杰,等 .AFLP分子标记构建中国对虾遗传连锁图谱的初步研究[J] .高技术通讯,2004,5:88-93.
[49] 岳志芹,王伟继,孔杰,等 .用AFLP方法分析中国对虾抗病选育群体的遗传变异[J] .水产学报,20.5.29(1):13-19.
[50] 张四明,分子生物学技术及其在渔业科学中的应用[J] .水产学报,1997,21(增刊):97-106.
[51] 张天时,刘萍,李健,等 .用微卫星DNA技术对中国对虾人工选育群体遗传多样性的研究[J] .水产学报,20.5.29(1):6-12.
[52] 张晓军,王兵,张绍萍,等 .中国对虾6种组织cDNA文库的构建[J] .海洋学报,20.5.27(5):92-95.
[53] 张煜,邓景耀 .渤黄海对虾标志放流实验[J] .海洋水产研究丛刊,1965,20:78-85
[54] 张志峰,马英杰,廖承义,等 .中国对虾幼体发育阶段的同工酶研究[J] .海洋学报,19.7.19(4):63-71.
[55] 真子渺,中岛国重,田川滕 .コウライエビの体长组成の変化について[J] .西水研报,1996,34:1-10.
[56] 真子渺,庄岛悦子 .标识放流によるコウライエビの移动と来游量の推定[J] .西水研报,1969,37:35-50.
[57] Amy L R,Danny L,Rushu L,et al. Genes dependent on zebraf ish cyclops function identif ied by AFLP differential gene expressionscreen[J]. Genesis,2000,26:86-97.
[58] Bachem C W B,Horvath B,Trindade L,et al. A potato tuber-expressedmRNA with homology tosteroid dehydrogenases affects gibberellin levels and plant development[J]. The Plant Journal,20.1.25(6):595-604.
[59] Bachem C W B,Oomen R J F J,Kuyt S,et al. Antisensesuppression of a potato α-SNAP homologue leads to alterations in cellular development and assimilate distribution[J]. Molecular Biology,2000,43:473-482.
[60] Bachem C W B,Van der H R S,de Bruijn S M,et al. Visualization of differential gene expression using a novelmethod of RNA f ingerprinting based on AFLP: analysis of gene expression during potato tuber development[J]. The Plant Journal,1996,9(5):745-753.
[61] Botstein D,White R L. Construction of gene linkagemap inman using restricion fragment length polymorphisms[J]. American Journal of Animal Gene,1980,32:314-331.
[62] Breyne P,Dreesen R,Cannoot B,et al. Quantitative cDNA-AFLP analysis for genomewide expressionstudies[J]. Molecular Genetics and Genomics,2003,269(2):173-179.
[63] Brown W M. Rapid Evolution of Animal Mitochondrial DNA Rapid Evolution of Animal Mitochondrial DNA[J]. Proceedings of the National Academy of Sciences,1979,(4):1967-1971.
[64] Cappelli K,Verini-Supplizi A,Capomaccio S,et al. Analysis of peripheral bloodmononuclear cells gene expression in endurance horses by cDNA-AFLP technique[J]. Research in Veterinary Science,20.7.82(3):335-343.
[65] Carr W,Sweenny J,Swingle J. The Oceanic Institute’s SPFshrimp breeding programstatus[A]. LSMSFP10th Anniverasry Review[C]. GCRL Sprcial Pulication,1994,(1):47-54.
[66] Dai J,Zhang Q,and Bao Z. Karyotypestudies on Penaeus orientalis[M]. J. Ocean. Univ. Qingdao,1989,19:97-103.
[67] Danzmann R G. Genetic discrimination of wild and hatchry populations of brook charr,Salvelinus fontinalis(Mitchll),in Ontario usingmitochondrial DNA analysis[J]. Journal of Fish Biology,1991,39:69-77.
[68] Durrant W E,Rowland O,Piedras P,et al. cDNA-AFLP reveals astriking overlap in race-specif ic resistance and wound response gene expression prof iles[J]. The Plant Cell,2000,12:963-977.
[69] Ekkapongpisit M,Wannatung T,Susantad T,et al. cDNA-AFLP analysis of differential gene expression in human hepatoma cells(HepG2)upon dengue virus infection[J]. Journal of Medical Virology,2007,79(5):552-561.
[70] Garcia D K,Dhar A K,Alcivar-Warren A. Molecular analysis of a RAPDmarker(B20)reveals twomicrosatellites and differentialmRNA expression in Penaeus vannamei[J]. Molecular Marine Biology and Biotechnology,19.6.5(1):71-83.
[71] Hedgecock D,Tracey M L,Nelson K. Genetic. In:Abele L G ed. The Biology of Crustacea,Vol.2. New York:Academic Press,1982,284-403.
[72] Lewontin R C. Amolecular approach to thestudy of genetic hetrozygosity in natural populations Ⅱ. Amount of variation and degree of hetrozygisity in natural populations of Drosphila pseudoobscura[J]. Genetics,1966,54:595-609.
[73] Li Z X,Li J,Wang Q Y,et al. AFLP-based genetic linkagemap of theshrimp Fenneropenaeus chinensis[J]. Aquaculture,2006,261:463-472.
[74] Lin J J,Kuo J. AFLP:A novel PCR-based assay for plant and bacterial DNA f ingerpint[J]. Focus,1995,17:52-56.
[75] Liu P,Meng X H,Kong J,et al. Polymorphic analysis ofmicrosatellite DNA in wild populations of Chineseshrimp(Fenneropenaeus chinensis)[J]. Aquactulture Research,2006,37(6):556-562.
[76] Liu Ping,Kong Jie,Shi Tuo et al. RAPD analysis of wildstock of Penaeidshrimp(Penaeus chinensis)in Chinese coastal waters of the Huanghai Sea and coastal waters of the Bohai Sea[J]. Acta Oceanologica Sinica,20.0.19(1):119-126.
[77] Marnik V,johan D P,Michiel J T E. AFLP-based transcript prof iling(cDNA-AFLP)for genome-wide expression analysis[J]. Nature Protocols,2007,2(6):1399-1413
[78] Moore S S,Whan V ,Davis G P et al. The development and application of geneticmarkers for the Kuruma prawn Penaeus japonicus[J]. Aquaculture,1999,173:19-32.
[79] Nei M,Koehn R Nei M. Genetic Distance between Populations[J]. American Naturalist,1972.1.6(949):219-223.
[80] K eds. Evolution of Gene and Proteins Sinauer,Sunderland[M].1983.147-164.
[81] Pruder G D. Healthshrimpsystemsseedsupply-theory and practice[A]//C L Browdy J S Hopkins eds. Swimming Throught Troubled Waters. Proc of thespecialsession onshrimp farming[C]. San Die go CA World Aquaculture Soc Baton Rouge LA,LSA.1995,40-52.
[82] Sugaya T,Jkeda M,Mori H et al. Inheritancemode ofmicrosatellites DNAmarkers and their use for kinship estimation in kuruma prawn Penaeus japonicus[J]. Fisheres Science,2002,68:299-305.
[83] Wang W J,Kong J,Bao Z M,et al. Isozyme variation in four populations of Penaeus Chinensisshrimp[J]. Chinese Biodiversity,2001,9:241-246.
[84] Waples R S. Genetic approaches to themanagement of Pacif icsalmon[J]. Fisheries,1990,15(5):19-25.
[85] Welsh J Petersen C M McClelland. Polymorphisms generated by arbitrarily primed PCR in themouse:application tostrain identif ication and genetic[J]. Nucleic Acids Research,1991,19(2):303-306.
[86] Williams J G,Kubelik A R,Livak K J,et al. DNA polymorphisms amplif ied by arbitrary primers are useful as geneticmarkers[J]. Nucleic Acids Research,1990,18(22):6531-6535.
[87] Wilson K,Li Y,Whan V,et al. Geneticmapping of the black tigershrimp Penaeusmonodon with amplif ied fragment length polymorphism[J]. Aquaculture,2002,204:297-309.
[88] Wolfus G M,Garcia D K A Alcivar-Warren. Application of themicrosatellite techniques for analyzing genetic diversity inshrimp breeding programs[J]. Aquaculture,1997,152:35-47.
[89] Wrgin I,Maceda L,Crittenden J. Use ofmitochondrial DNA polymorphisms to estimate the relative contribution of Hudson River and Chespeake Baystriped bassstocks to themixed f ishery on the Atlant coast[J]. Transactions of the American Fisheries Society,1993,122:669-684.
免责声明:以上内容源自网络,版权归原作者所有,如有侵犯您的原创版权请告知,我们将尽快删除相关内容。
















