海洋微生物检测中常常需要进行某类群微生物的检测,比如病原菌、病毒或是涉及海洋生物制品安全的微生物等。检测这类微生物需要通过特异性强、准确度高的检测技术手段,在现在的检测方法中包括传统分类鉴定技术、基于抗原抗体反应的血清学方法以及基于特异性的基因检测的分子生物学方法等。
1.传统分类鉴定技术
常规宏观菌落形态学观察,主要观察菌落在其适合的培养基上的生长状况:细菌在固体培养基上的生长形态、大小、颜色是否均匀一致、菌落个体表面及边缘的生长状况;在液体培养基上的浑浊状况、沉淀状况、液面菌膜状况;半固体培养基上穿刺接种后,观察细菌是否沿着接种线生长以及其生长状态是呈毛刷样生长还是均匀生长,上下生长是否一致;在鉴别培养基上培养,观察结果是否跟预期结果相同。
细菌的个体形态学观察,即通过显微镜观察。观察前细菌需要着色,要根据预先确定的观察项目选择与之相对应的染色方法,目前常用的染色方法包括革兰氏染色法、美蓝染色法、Ziehl-Neelsen染色法、姬姆萨染色法、鞭毛染色法、芽孢染色法等。尽量挑选对数生长期的细菌进行染色观察,此时的细菌生长处于幼期,利于观察,因为一些陈旧细菌的染色结果会有变化,如本为革兰氏阳性菌经过长期搁置后染色结果可能为革兰氏阴性。同时细菌培养时要注意培养基的挑选,一些细菌的特殊结构如荚膜、鞭毛、菌毛、芽孢等需要在特定的培养基上才能正常发育;有的细菌如炭疽在一般的培养基上不形成荚膜,只有在动物体内才能形成明显的荚膜,所以需先接种实验动物后用病料进行涂片镜检。在镜检时观察细菌基本形态结构和大小及其排列状态、菌端形状、有无两极染色、有无形成芽孢和荚膜等。
形态学鉴定辅以生化试验在细菌鉴定中具有重要的意义,生化试验是根据细菌培养过程中不同菌种所产生的新陈代谢产物不同,表现出不同的生长特性。通过生物化学的方法来检测这些物质的存在与否,从而能够得到细菌的鉴定结果。如糖(醇)类代谢试验、氨基酸和蛋白质代谢试验、有机酸盐和胺盐利用试验、呼吸酶类试验、毒性酶类试验等。
血清型鉴定是根据细菌具有相对特异性的抗原结构这个特点进行微生物鉴定的特异方法。抗原的特异性程度又存在于属间细菌所共有的共同抗原,这种抗原的存在能表明其属性。另一类特异性抗原只存在于特定的种、型,是确定细菌种、型的重要依据。通过专门的分型血清即可对这些细菌的血清型进行鉴定。
细菌毒力的测定,病原菌侵入机体能否引起疾病与其本身的毒力、侵入机体的数量和侵入部位有关。不同的病原菌或同种细菌不同的型或株,毒力常不一致。常用LD50或ID50表示毒力,即在一定时间内,能使一定条件的某种动物半数死亡或感染需要的最小细菌数或毒素量。毒力测定在菌种鉴定过程中可用于鉴别一些有代表性的菌株。LD50可用Reed-Muench公式进行计算。
2.血清学检测技术
血清学检测技术是基于免疫学中抗原同抗体能特异性结合,由此可通过抗原/抗体来检测抗体 / 抗原的检测技术。
酶联免疫吸附试验(enzyme-linked immunosorbent assay,ELISA)是以免疫学反应为基础,将抗原、抗体的特异性反应与酶对底物的高效催化作用相结合起来的一种敏感性很高的试验技术。试验主要包括抗原或抗体的固相化以及抗原或抗体的酶标记,结果通过酶活性测定来确定抗原或抗体的含量。将酶分子与抗体或抗抗体分子共价结合,此种结合要求既不改变抗体的免疫反应活性,也不影响酶的生物化学活性。用于标记抗体的酶很多,常用的有辣根过氧化物酶(horseradish peroxidase,HRP)、碱性磷酸酶(alkaline phosphatase,AP)等。标记方法有氧化法与交联法。氧化法常采用过碘酸钠,故又称过碘酸钠法。过碘酸钠是一种强氧化剂,能将酶的甘露糖部分(如HRP中与酶活性无关的部分)的羟基氧化成醛基,然后与抗体的氨基结合,形成酶标抗体。交联法常用的交联剂是戊二醛,故又称戊二醛法,主要利用戊二醛分子上对称的两个醛基,分别与酶和蛋白质分子中游离的氨基、酚基等以共价键结合而进行标记。
免疫传感器就是利用抗原(抗体)对抗体(抗原)的识别功能而研制成的生物传感器。免疫传感器使用光敏元件作为信息转换器,生物识别分子被固化在传感器,通过与光学器件的光的相互作用,产生变化的光学信号,通过检测变化的光学信号来检测免疫反应。免疫传感器作为一种新兴的生物传感器,以其鉴定物质的高度特异性、敏感性和稳定性受到青睐,将传统的免疫测试和生物传感技术融为一体,不仅减少了分析时间、提高了灵敏度和测试精度,也使得测定过程变得简单,易于实现自动化,有着广阔的应用前景。
3.分子检测技术
分子检测技术是现今进行病原检测和诊断的主要技术方法,其根据目标生物的一段特异性的基因序列,进行序列扩增或是特异性探针结合,通过电泳或是显色或是荧光等方式进行阳性结果判定。
聚合酶链式反应(polymerase chain reaction,PCR)是体外酶促合成特异DNA片段的一种方法,由高温变性、低温退火及适温延伸等几步反应组成一个周期,循环进行,使目的DNA得以迅速扩增,具有特异性强、灵敏度高、操作简便、省时等特点。它不仅可用于基因分离、克隆和核酸序列分析等基础研究,还可用于疾病的诊断或任何有DNA、RNA的地方,因此又称无细胞分子克隆或特异性DNA序列体外引物定向酶促扩增技术。该技术由美国科学家PE(Perkin Elmer,珀金·埃尔默)公司遗传部的穆利斯发明,由于PCR技术在理论和应用上的跨时代意义,穆利斯因此获得了1993年诺贝尔化学奖。
PCR技术的基本原理类似于DNA的天然复制过程,其特异性依赖于与靶序列两端互补的寡核苷酸引物(图2-1)。PCR由变性、退火、延伸三个基本反应步骤构成:① 模板DNA的变性:模板DNA经加热至93℃左右一定时间后,使模板DNA双链或经PCR扩增形成的双链DNA解离,使之成为单链,以便它与引物结合,为下轮反应作准备;② 模板DNA与引物的退火(复性):模板DNA经加热变性成单链后,温度降至55℃左右,引物与模板DNA单链的互补序列配对结合;③ 引物的延伸:DNA模板 — 引物结合物在TaqDNA聚合酶的作用下,以dNTP为反应原料,靶序列为模板,按碱基互补配对与半保留复制原理,合成一条新的与模板DNA链互补的半保留复制链,重复循环变性 — 退火 — 延伸三过程就可获得更多的“半保留复制链”,而且这种新链又可成为下次循环的模板。每完成一个循环需2~4min,2~3h就能将待扩目的基因扩增放大几百万倍。
PCR的模板可以是DNA,也可以是RNA。模板的取材主要依据PCR的扩增对象,可以是病原体标本如病毒、细菌、真菌等,也可以是病理生理标本如细胞、血液、羊水细胞等,法医学标本有血斑、精斑、毛发等。标本处理的基本要求是除去杂质,并部分纯化标本中的核酸。多数样品需要经过SDS和蛋白酶K处理。难以破碎的细菌,可用溶菌酶加EDTA处理。所得到的粗制DNA,经酚、氯仿抽提纯化,再用乙醇沉淀后用作PCR反应模板。
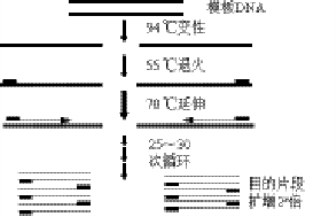
图2-1 PCR反应原理示意图
实时定量PCR(Real-time PCR)是指在PCR指数扩增期间,通过连续检测荧光信号强弱的变化来即时测定特异性产物的量,并根据此推断目的基因的初始量。该技术实现了PCR从定性到定量的飞跃,使得临床检验结果更具有精确性。目前实时定量PCR作为一个极有效的实验方法,已被广泛地应用于分子生物学研究的各个领域,在微生物的检测方面具有很好的应用前景和研究价值。
2000年日本学者Notomi在《核酸研究》杂志上公布了一种新的基因诊断技术,即LAMP(Loop-mediated isothermal amplification),中文名为“环介导等温扩增反应”,受到了世界卫生组织(WHO)各国学者和相关政府部门的关注,短短几年,该技术已成功地应用于SARS、禽流感、HIV等疾病的检测中。LAMP法不需要长时间的温度扩增,只需在恒温条件下作用1h,即可将极微量的核酸物质扩增至109的拷贝数,是一种经济、简便、灵敏、特异的核酸扩增方法。LAMP反应的结果可直接靠扩增副产物焦磷酸镁的沉淀浊度进行判断,亦可通过加入荧光染料,肉眼观察荧光的强弱来判断扩增结果,适合现场或是基层的快速检测。而对LAMP扩增产物进行琼脂糖凝胶电泳分析时,在紫外灯下可观察到典型的梯状条带,可通过不同梯形来区分特异性扩增与非特异扩增。
此外,利用分子生物学原理建立的微生物检测和鉴定技术还包括斑点杂交(dot blot)、原位杂交(in situ hybridization)、基因芯片(gene chip)技术等。例如,国外一些学者用基因芯片技术进行了海洋微生物检测的研究。Taroncher-Oldenburg等(2003)利用70mer长的寡核苷酸作为探针来检测河水、海湾等环境中氮循环基因的多样性,这些基因包括amoA,nifH,nirK和nirS等。他们用PCR扩增这些基因的全长片段并同时进行荧光标记。研究表明,每种对象的检测灵敏度为10pg DNA,大约是107拷贝数。Tiquia等(2006)利用763个50mer长的寡核苷酸探针来检测美国普基特海湾(Puget Sound)不同深度的沉积物中amoA,pmoA,nirS,nirK,nifH和dsrAB基因的分布情况,这些基因是编码生态系统中硝化作用、甲烷氧化作用、反硝化作用、固氮作用以及硫还原作用(sulfur reduction)中的关键酶。利用该基因芯片技术,发现这些基因的多样性在深的沉积物中明显比在浅的沉积物中低。
4.快速鉴定系统和自动化分析
由于传统鉴定方法需从形态、生理生化特征等方面进行数十项试验,才能将细菌鉴定到种,工作量大,花费时间长。因此,20世纪70年代起国外开始实行成套的标准化鉴定系统和与之相结合的计算机辅助鉴定软件,使细菌鉴定技术日益朝着简便化、标准化和自动化的方向发展。目前常见的细菌鉴定系统有API系统、BIOLOG系统、MIS(MIDI)系统、Enterrotube系统、PhP系统、VITEK系统等。这些简单鉴定系统的设计,最初是针对肠杆菌科的细菌及相关的G-杆菌,目的在于简化细菌鉴定的生化项目。简易细菌鉴定系统对临床上分离菌株的鉴定,取得了较满意的效果,准确率高、操作简便,并能大大缩短细菌鉴定时间。然而,对于从海洋环境中分离到的细菌,采用这些系统尚有一定的局限性,往往不能将所鉴定的菌株鉴定到种,而且有些鉴定结果不可靠。这主要是由于海洋细菌所要求的生长条件和常规的肠杆菌科的不同,另外,这些快速鉴定系统所收集的标准菌株数据库中尚无足够的海洋细菌的特征资料。比如,MacDonell等报道用API20E鉴定海洋及河口细菌时,稀释液的盐度对鉴定结果会产生影响。因此,对于这些系统在海洋细菌鉴定中的应用,有待于做进一步的试验比较,积累更多的资料。对其中的某些试验条件,如温度和培养基盐度等应作适当的调整,使之更适合于海洋细菌的鉴定。下面对几种常见细菌快速鉴定系统作一简单介绍。
(1)API细菌数值鉴定系统。
API系统是目前世界上应用最为广泛的细菌鉴定系统,主要由含20种脱水基质的微型管、API试验条以及试验结果读数表和检测试剂等组成。每个试验条可对1株细菌进行23项生化试验。挑取分离纯化后的单个菌落,制成菌悬液,按要求加入API试验条的微型管中,适宜温度下培养24h。以其自身代谢产物颜色的变化或加入试剂后颜色的变化加以鉴定。其结果以一个7位数字形式查对检索表或直接输入计算机即可得到相应的种名。其中API20E主要用于检测发酵型G- 杆菌,可对98种肠道G- 杆菌进行准确鉴定。
(2)Enterotube系统。
Enterotube系统是用无菌的互相间隔开的塑料管组成一个整体,其中8个小室,分别装有不同酶作用物和指示剂,每个塑料管两端各有一个帽盖并封存一只接种针,接种时除去接种针外端的帽盖,并用该针从原始培养平皿上挑取菌落,然后伸入进塑料管中,将菌种接种到培养基上,再将针体部分抽出,拆掉留在管外的部分。遗留在管内的部分针体有助于保持厌氧条件,接种针抽出部分则可提供需氧条件。盖上帽盖,将接种管孵育过夜。该装置可做下列试验:葡萄糖产酸产气、赖氨酸和鸟氨酸脱羧酶、硫化氢、靛基氢、乳糖和卫矛醇发酵、苯丙氨酸脱氢酶、尿素酶和柠檬酸盐等试验。改进后的Enterotube型,除可做上述11种试验外,又增加了侧金盏花醇、阿拉伯糖、山梨醇和VP试验4种。
(3)Biolog全自动或手动细菌鉴定系统。
Biolog微生物自动分析系统是美国Biolog公司从1989年开始推出的一套微生物鉴定系统,该系统主要根据细菌对糖、醇、酸、醋、胺和大分子聚合物等95种碳源的利用情况进行鉴定。细菌利用碳源进行呼吸时,会将四哇类氧化还原染色剂(TV)从无色还原成紫色,从而在鉴定微平板(96孔板)上形成该菌株特征性的反应模式或“指纹图谱”,通过纤维光学读取设备 — 读数仪来读取颜色变化(分为“自动”和“人工”两种读取方式),由计算机通过概率最大模拟法将该反应模式或“指纹图谱”与数据库相比较,可以在瞬间得到鉴定结果,确定所分析的菌株的属名或种名。以Biolog系统6.01版数据库为例,包含了革兰氏阴性好氧菌524种、革兰氏阳性好氧菌341种、厌氧菌361种、酵母菌267种、丝状真菌(含部分酵母菌)619种以及几种特殊的病原菌,目前这个数据库已进行更新。利用微生物快速系统进行细菌鉴定,能节省时间,但由于其在数据比对过程中记入一些非特异性的阳性反应孔,会造成偶然误差,从而引起个别鉴定结果不稳定,另外此类仪器本身及其耗材价格均较高。
(4)Sherlock MIS系统。
Sherlock MIS系统是由美国 MIDI 公司开发,专用于微生物鉴定。PLFAs的组成和含量水平具有种属的特异性,Sherlock MIS系统需通过气相色谱分析。Sherlock MIS系统菌种库包括好氧菌1500种,厌氧菌800种及酵母菌(包括放线菌)30000种。分离纯化后的细菌,按规定方法提取脂类物质,注入仪器的层析柱中,仪器可自动分析细菌样品中的脂类物质成分及含量,并与计算机内存储的细菌资料进行比较,鉴定出细菌的种名。
(5)RiboPrinter鉴定系统。
RiboPrinter全自动微生物基因指纹鉴定系统,是迄今为止全球唯一自动化DNA指纹仪器,用于鉴定、鉴别微生物并给出分子信息。该系统为环境分离物、病原菌、有害微生物、质控菌株、有益微生物等建立了一个全自动的基因指纹图库,这一精确到菌株水平的鉴定具有追溯细菌来源、对所有微生物环境进行控制及流行病学调查等功能。RiboPrinter鉴定系统是基于核糖体DNA分型技术,使用限制性内切酶消化待检菌的DNA,消化后的基因片段经电泳分离转印到硝酸纤维素膜上,与DNA探针杂交,该探针包括编码高度保守的16S rDNA和23S rDNA片段以及间隔序列,杂交信号经处理产生“条形码”状条带图像(RiboPrinter模式),即rDNA基因指纹图谱,并与数据库比较从而达到细菌鉴定、分型的目的。该系统目前可以区分超过180个属的1400多株菌。
免责声明:以上内容源自网络,版权归原作者所有,如有侵犯您的原创版权请告知,我们将尽快删除相关内容。

















