实验二十六 酿酒酵母乙醇脱氢酶ADH3基因敲除
一、实验目的
(1)学习基因敲除在基因功能研究和酵母育种中的应用。
(2)掌握Cre/loxP系统介导的基因敲除的原理及操作过程。
二、实验原理
基因敲除技术在基因功能研究和微生物育种中发挥重要作用。该技术主要是将构建好的基因敲除序列组件转化至细胞,由同源重组实现目标基因的敲除。其中的基因敲除序列组件是在抗性基因的两侧分别连接一段与目标基因两侧同源的短片段。同源重组的效率在很大程度上依赖于基因敲除组件的同源片段的长度,在酵母实验中一般使用的同源序列为35~45bp。目前常用的基因敲除系统有Cre/loxP、Flip/FRT和TELEN等。
Cre/loxP系统由来自E.coli噬菌体P的Cre重组酶和由2个13bp的反向重复序列和1个8bp的间隔区域构成的loxP位点两部分组成(如图26-1)。Cre是1 个38kDa的重组酶蛋白,它可以介导loxP的34bp重复序列的位点特异性重组,切除同向重复的2个loxP位点的DNA片段和1个loxP位点,保留1个loxP位点。Cre酶活性具有可诱导的特点,将Cre基因置于可诱导的启动子控制下,通过诱导表达Cre重组酶而将loxP位点之间的基因切除,可实现特定基因在特定时间或者组织中的失活。因此Cre/loxP系统是进行基因组定点突变、研究基因精细结构的新途径。
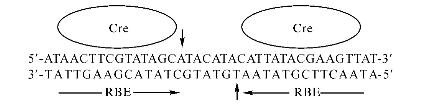
图26-1 loxP位点序列
三、实验材料
(一)菌种及质粒
(1)菌种:酿酒酵母(Saccharomyces cerevisiae)BY4741,大肠杆菌DH5α。
(2)质粒:pUG6、pSH65,其中pUG6质粒带有Kanr抗性基因,两端具有loxP位点,pSH65质粒带有Zeocin筛选标记,在半乳糖诱导下表达Cre酶。
(二)培养基
(1)YPD培养基:酵母提取物1%,蛋白胨2%,葡萄糖2%,121℃灭菌20min,用于酵母菌的活化与培养。
(2)麦芽汁培养基:麦芽和水以1∶4的比例混合,60℃水浴4h,8层纱布过滤,滤出液108℃灭菌20min,用于酵母菌发酵培养实验。
(三)主要工具酶和试剂
ExTaq DNA聚合酶,rTaq DNA聚合酶,内切酶(HindⅢ、SalⅠ、BglⅡ),pMD18-T载体试剂盒,胶回收试剂盒,G418、Zeocin和氨苄青霉素。其他试剂均为国产分析纯试剂。
(四)引物设计
敲出组件的引物L1、L2是依据酿酒酵母ADH3基因和pUG6质粒中loxPKan-loxP设计的L1、L2的5′端分别有与酵母基因组ADH3基因起始密码子(包括)前和终止密码子(包括)后相同的45个碱基,用来进行同源重组。L1(L2)的3′端有19(22)个碱基与质粒pUG6中loxP-Kan-loxP两侧的上游(下游)某段序列相同,用来扩增loxP-Kan-loxP。
引物A、B、C、D是依据酵母基因组ADH3基因设计的,用来验证基因组中是否含有ADH3基因序列,A位于ADH3基因ORF的上游5区域,B和C位于ORF上,D位于ADH3基因ORF的下游3区域。引物KB和KC是依据敲除组件L1-L2的loxP-Kan-loxP设计的,位于Kanr基因上,用于验证敲除组件是否正确重组到原ADH3位置。这6条引物相互配对使用,用于敲除过程的验证,引物对分别为:A-B、C-D、A-D、A-KB、KC-D。
表26-1 引物序列

(五)仪器与设备
PCR仪、核酸电泳仪、凝胶成像分析仪、冷冻离心机、恒温培养箱、摇床、水浴锅等。
四、实验步骤
(一)酿酒酵母总DNA的提取
取1.5mL新培养的酵母培养液,经离心收集酵母细胞,提取酵母总DNA,并琼脂糖氧胶电泳验证。
(二)PCR获得敲除组件L1-L2
以L1、L2为上下游引物,pUG6为模板进行PCR扩增,获得扩增产物L1-L2。PCR标准扩增反应程序为:94℃热启动5min,94℃变性30s,55℃退火30s,72℃延伸2min共30个循环,最后72℃延伸10min。取3μL PCR扩增产物做琼脂糖凝胶电泳验证。
(三)乙醇沉淀扩增产物L1-L2
通过测序并比对,如果PCR方法获得的敲除组件准确无误,取200μL PCR扩增产物至无菌干净的1.5mL离心管中,加入2.5倍体积的冰冷无水乙醇。混匀,置-20℃冰箱中沉淀1h以上。10 000rpm,4℃离心10min,去上清,用体积分数70%的乙醇洗2次,室温下10 000rpm离心3min。除去乙醇,晾干,用50μL无菌ddH2O溶解沉淀的DNA,-20℃冰箱保藏,留做醋酸锂法转化时使用。
(四)转化
采用醋酸锂高效化学转化法转化敲除组件。
(1)感受态细胞制备:用无菌接种环取冻存的酵母菌,在YPD平板上连续划线接种,3d后在YPD平板上挑取单个菌落接种在10mL YPD培养基中摇菌过夜。把10mL菌液转移到100mL YPD培养基中振荡培养至A600为0.6。将菌液置于灭菌的离心管中离心,弃上清液。无菌水重新悬浮细胞,再离心收集菌体。于离心管中加入1×TE250μL,1×LiAC250μL,制成感受态细胞,需现做现用。
(2)重组线性DNA片段转化到酵母细胞:煮沸10mg/mL鲑鱼精DNA 5min,迅速置于冰上。取无菌1.5mL EP管依次加入线性化的DNA 20μL,鲑鱼精DNA 10μL,感受态细胞200μL,用枪头混匀后,依次加入10×TE 60μL,10× LiAC 60μL,50%PEG4000 480μL,用枪头混匀,30℃摇菌30min,加入70μL的DMSO,轻轻颠倒2~3次,置于冰42℃水浴锅中热休克15min,冰上放置2min,离心弃上清液,加入200μL 1×TE悬浮,制成菌悬液。
(五)筛选敲除菌株
转化后的酵母菌悬液涂布到含有200μg/mL G418的YPD平板上,28℃培养2~3d。通过影印平板法筛选转化子,那些既能在不含任何抗生素的YPD上生长又能在含有G418的YPD平板上生长的菌落可能是正确重组的敲除组件L1-L2的菌落。
(六)PCR验证转化子
挑取转化子菌落,提取基因组DNA,分别用引物对A-B、C-D、A-D、A-KB、KCD和L1-L2进行PCR验证,将PCR扩增产物做琼脂糖凝胶电泳验证。
(七)去除抗性标记
采用醋酸锂法将质粒pSH65(带有zeocin抗性标记)分别转入不同菌株的阳性克隆子,YPG培养基培养过夜,使之在半乳糖的诱导下表达Cre酶切除阳性克隆子染色体上的G418筛选标记。培养物稀释涂布于YPD平板,待菌落长出后,通过影印法将细胞转移到两份YPD平板中,一份含50μg/mL zeocin,一份含250μg/mL G418,30℃培养2~3d后观察,zeocin平板上长出,但G418平板上不生长的菌落即为G418抗性标记已去除的转化子。转化子在YPD培养基中连续传代培养直至丢失pSH65质粒,得到不含筛选标记的转化子菌株。
(八)乙醇脱氢酶的酶活检测
将验证乙醇脱氢酶Ⅲ缺失的酿酒酵母菌株接种到麦芽汁培养基中,30℃,180r/min培养48h,离心收集菌体。用超声破碎法获得粗酶液,每隔15s测定340nm波长下NADH的吸光值。酶活定义:单位湿细胞在单位时间使吸光值每产生0.001的改变定义为一个酶活。
五、实验结果记录
(1)琼脂糖凝胶电泳PCR产物,求出DNA长度,判断PCR产物是否是目标产物?
(2)挑取的菌落中是否有假阳性克隆?
六、思考题
(1)基因敲除的方法有哪些?它们的原理分别是什么?
(2)在引物A1和A2的5′端分别设计41bp和40bp与酿酒酵母ADH3基因外侧序列相同碱基的目的是什么?
[1]宋浩雷,郭晓贤,杨月梅,等.酿酒酵母ADH3基因的敲除[J].工业微生物,2006,36(4):28—32.
[2]和东芹,肖冬光,吕烨.Cre/loxP系统介导的基因重组及其在酵母中的应用[J].中国生物工程杂志,2008,28(6):46—49.
[3]蒋咏梅.微生物育种学实验[M].北京:科学出版社,2012.
免责声明:以上内容源自网络,版权归原作者所有,如有侵犯您的原创版权请告知,我们将尽快删除相关内容。















