我国在医院信息化的快速发展过程中,医院信息系统(HIS)、社区卫生服务系统(CHSS)等不断被开发和应用。这些医学信息系统往往由不同的医疗机构自行开发或采购,执行的是不同的私有数据标准,积累了大量来自于不同系统、不同机构的异构数据资源。由于数据结构标准和数据管理标准的差异,现有医学信息系统的互联互通存在重重障碍,信息孤岛问题十分突出,这使得个人健康信息在各医疗机构之间不能方便及时地互相交换,医疗数据重复利用率低,导致重复检查、重复化验等不必要的医疗资源浪费,且进一步加剧了“看病贵、看病难”的现状。
(一)数据标准化
1.医疗数据相关国际标准
(1)医疗健康信息传输与交换标准HL7。目前,国际上最具影响力并被广泛使用的医疗系统标准是由美国非营利性机构Health Level Seven Inc.研究开发的HL7(Health Level Seven),它是基于消息实现数据传递的标准规范,可以应用于多种操作系统和硬件环境,也可以进行多应用系统间的文件和数据交换。它是医疗领域不同应用系统之间电子数据传输的协议,主要目的是要发展各型医疗信息系统间,如临床、检验、管理及行政等各类医疗数据交换的标准。
美国政府规定国内全部医院信息系统在2004年5月以后必须支持HL7,该规定得到了绝大部分医疗机构和医疗器械厂商的积极响应,HL7已经成为美国政府强制执行的标准。目前,HL7组织已经吸纳了美国以外的世界上几十个国家及地区,包括中国、澳大利亚、新西兰、德国、荷兰、英国、捷克、立陶宛、阿根廷、加拿大、芬兰、土耳其、瑞士、韩国、日本、印度、南非以及中国台湾地区等。
HL7在1987年7月份正式颁布了1.0版本,到今天它的版本已经发展到3.0版,HL7 3.0版本是一套在2.x版本基础上发展起来的新的标准体系,目前仍处于发展和完善的过程中。HL7标准所涉及的内容也由最初定义的病患信息、住院、出院、转院和医嘱录入等少数几方面内容,发展到今天整个医疗业务活动的方方面面。
HL7是建立在开放系统互连标准第七层(应用层)的应用标准,针对HIS的应用要求,HL7建立了HIS的基本应用模型,采用消息传送机制实现不同应用系统之间的数据传输和功能调用。
①消息结构:在HL7协议中,消息(Message)是各应用系统之间数据交换的基本单元,它是系统间传输数据的最小单位,每个消息都是用一个消息类型来表示其用途。一个消息由一组规定次序的多个段(Segment)组成,每个段都有相应的名称。一个消息中的第一个段总是消息头段(Message head segment),它指明了发送和接收的程序名称、消息类型、唯一的消息ID号等。而一个段又由多个数据字段(Data Field)组成,段是数据字段的一个逻辑组合,用一个唯一的三字符代码所标志,这个代码称作段标志。
②HL7接口引擎:HL7接口引擎由发送/接收模块(Send/Receive module)、转换模块(HL7 Adaptor module)、应用接口模块(HL7 API module)、HL7资源模块(HL7 Resource module)和对照模块(Mapping module)等几部分组成。
发送/接收模块:支持TCP/IP通讯协议,医疗系统向数据中心发送符合HL7标准的医疗数据信息。数据中心接收并解析信息,将解析后的信息存到数据中心的数据库中,完成后回复发送端一个ACK确认信息,确认信息已经发送成功。
转换模块:检查验证发送/接收医疗数据信息格式的正确性和完整性,实现字符串格式数据与XML格式之间的相互转换。
应用接口模块:提供符合HL7标准的应用接口,医疗应用系统可以通过调用应用接口函数,并按照HL7标准格式填写参数,实现向其应用系统发送数据。
HL7资源模块:对各类实际应用的HL7医疗信息事件提供支持。
对照模块:可以按照应用系统的需要进行定制,提供翻译对照功能。
(2)医学数字成像和通信标准DICOM。DICOM(Digital Imaging and Communications in Medicine)即医学数字成像和通信,是医学图像和相关信息的国际标准,它定义了质量能满足临床需要且可用于数据交换的医学图像格式,用于解决不同生产厂商不同型号的设备产生的图像格式不一致、不同设备之间信息资源难以互用的问题。随着以CT为代表的数字成像诊断设备在临床得到广泛应用,美国放射学院(ACR)和国家电气制造协会(NEMA)在1983年成立了一个专门委员会,开始制定医学图像存储和通信标准,以达到以下目的:推动不同制造商的设备之间数字图像信息通信标准的建立;促进和扩展图片归档及通讯系统(PACS),使它可以与其他医院信息系统进行交互;允许广泛分布于不同地理位置的诊断设备创建统一的诊断信息数据库。
ACR-NEMA联合委员会于1985年发布了最初的ACR-NEMA1.0版本,又分别于1986年10月和1988年1月发布了两个校订版本。1988年该委员会推出ACR-NEMA 2.0版本,由于技术不成熟,这些版本没有得到广泛采用。到1993年发布的DICOM 3.0标准,是在ACR-NEMA 2.0版本的基础上,增加了通信方面的规范,并且重新修改了图像格式中部分信息的定义,目前,DICOM 3.0标准已发展成为医学影像信息学领域的国际通用标准。
①DICOM数据结构和文件格式:DICOM标准的数据结构在它的第五部分进行了介绍,定义了数据集(Data Set)来保存信息对象定义(IOD)。这里,IOD是信息实体的抽象,是DICOM命令的作用受体。数据集又由多个数据元素(Data Element)组成,每个数据元素描述一条信息,如患者姓名、图像类型等。DICOM数据元素由八位十六进制数的标记唯一确定,如八位十六进制数“0008,0016”,前四位表示组号,后四位表示元素号。DICOM数据元素分为两种,即:
标准(Standard)数据元素,组号为偶数,含义在标准中已定义。
私有(Private)数据元素,组号为奇数,其描述信息的内容由用户定义。
②DICOM标准组成:DICOM标准由多文档组成,共分为以下18部分。
第一部分,引言和综述。简要介绍了DICOM标准的概念及组成,并简要介绍了其他部分的内容。
第二部分,兼容性。详细定义了DICOM的兼容性概念,要求制造商精确地描述其产品的DICOM兼容性,即构造一个该产品的DICOM兼容性声明,它包括选择的信息对象、服务类、消息传递、数据编码方法等。
第三部分,信息对象。此部分定义了信息对象和信息对象类,包括普通信息对象和复合信息对象。许多信息对象定义中有若干组属性是类似的,这些组被集中在一起形成了一系列多个信息对象定义都可以使用的公共模块。
第四部分,服务类规范。定义了一些服务类,服务类详述了作用于信息对象上的命令及其产生的结果。
第五部分,数据结构及编码。描述了怎样对信息对象类和服务类进行构造和编码。
第六部分,数据字典。数据字典是所有表达信息的DICOM数据元素的集合,它定义了数据元素及其标识符、值类型、数据类型等。
第七部分,信息交换。定义了DICOM命令的结构,以及进行信息交换通讯的医学图像应用实体所用到的服务和协议。
第八部分,信息交换的网络通讯支持。说明了在网络环境下DICOM如何支持TCP/IP协议和ISO-OSI协议。
第九部分,信息交换的点对点通讯支持。说明了与 ACR-NEMA 2.0兼容的点对点通讯的服务和协议。
第十、十一、十二部分,定义了医学影像信息的存储媒体和数据交换的文件格式。
第十三部分,打印管理的对点对点通讯支持,它描述了打印提供者在点对点连接情况下支持DICOM打印所需的服务和协议。
第十四部分,灰度图像显示函数,它描述了灰度图像显示的标准函数,说明了如何调整灰度图像和显示系统。
第十五部分,安全性和系统管理,它定义了DICOM安全模型。
第十六部分,绘制资源目录。
第十七部分,信息解释。
第十八部分,Web获取DICOM永久对象。
(3)国际疾病分类标准编码ICD-10。国际疾病分类(International Classification of Diseases,ICD),是世界卫生组织制定的国际统一的疾病分类方法,它根据疾病的病因、病理、临床表现和解剖位置等特性,将疾病分门别类,并用编码的方法来表示的系统。目前,全世界通用的是第10次修订本《疾病和有关健康问题的国际统计分类》(ICD-10)。
ICD主要依据病因、部位、病理及临床表现(包括:症状体征、分期、分型、性别、年龄、急慢性发病时间等)4个主要特征对疾病进行分类。ICD疾病分类的基础是对疾病的命名,没有名称就无法分类。疾病又是根据它的内在本质或外部表现来进行命名,因此疾病分类的依据就是疾病的本质和表现,分类与命名之间存在一种对应关系。当对于某个疾病名称赋予一个唯一编码时,这个编码就表示了该疾病的本质和表现。
疾病分类编码的操作方法包括4个步骤:首先要确定主导词,相当于在图书馆中检索时所用的主导词。确定主导词后,在第三卷字母索引中查找编码。再把查到的编码在第一卷类目表中核对编码。由于肿瘤要求有两个编码,因此,需要重新操作一遍。
(4)观测指标标识符逻辑命名与编码系统LOINC。观测指标标识符逻辑命名与编码系统(Logical Observation Identifiers Names and Codes,LOINC)由LOINC委员会及Regenstrief医疗保健研究院负责其开发和维护工作,LOINC数据库旨在促进临床观测指标结果的交换与共享。其中,LOINC术语涉及用于临床医疗护理、结局管理和临床研究等目的的各种临床观测指标,如血红蛋白、血清钾、各种生命体征等。
LOINC数据库实验室部分所收录的术语涵盖了化学、血液学、血清学、微生物学(包括寄生虫学和病毒学)及毒理学等常见类别或领域;还有与药物相关的检测指标,以及在全血计数或脑脊髓液细胞计数中的细胞计数指标等类别的术语。LOINC数据库临床部分的术语则包括生命体征、血液动力学、液体的摄入与排出、心电图、产科超声、心脏回波、泌尿道成像、胃镜检查、呼吸机管理、精选调查问卷及其他领域的多类临床观测指标。
2.国内卫生信息数据标准化建设 2006年,国家卫生部组建了卫生部卫生标准委员会卫生信息标准专业委员会,负责卫生数据标准的研究、起草、制定和推广应用工作。在卫生数据标准化的研究与数据标准推广应用过程中,数据集这一新的医疗数据组织方式在我国得到了应用和推广。数据集是具有一定主题、可以被标识并能够被计算机处理的数据集合,是一种基于主题进行数据资源收集与组织的新型数据组织方式和数据资源的“封装”单元。2012年,卫生部颁布了《卫生信息基本数据集》、《卫生信息数据元目录》、《卫生信息数据元值域代码》,其中,《卫生信息基本数据集》主要由《基本信息数据集》、《疾病管理基本数据集》、《疾病控制基本数据集》、《卫生管理基本数据集》、《医疗服务基本数据集》等几部分构成,为解决不同领域、不同层次的医疗卫生数据标准化奠定了基础。
(1)数据集与卫生信息数据集
①数据集:数据集是具有主题的、可标识的、能被计算机处理的数据集合。
主题:围绕着某一项特定任务或活动进行数据规划和设计时,对其内容进行的系统归纳和描述。数据集主题具有划分性和层级性,划分性即对主题进行分门别类,主题间通过不同的命名,将相同属性的主题归并在一起形成相同的类;层级性是指主题可被划分成若干子主题或子子主题。
可标识:即可以用规范的名称和标识符对数据集进行标记,以供识别。
能被计算机处理:即可以通过计算机技术(软硬件、网络),对数据集内容进行发布、交换、管理和查询应用。
数据集合:即按照数据元所形成的若干数据记录所构成的集合。
②卫生信息数据集:卫生信息数据集是在医疗卫生领域,为满足政府卫生决策、业务处理、科学研究、信息发布与绩效评价等需求,按照数据集概念设计、归纳、整合的主题信息集合。卫生信息数据集主要分为业务系统建设类的基本数据集、信息发布类统计数据集、为满足特定目的收集整理制作的数据集三种类型。
业务系统建设类的基本数据集:包括医疗、公共卫生、卫生监督等领域为了满足业务信息系统规范化建设和领域内部以及领域间数据交换与共享需求,设计归纳的各系统或功能模块所包含的最小数据元素的集合。如个人健康档案、住院患者入出转、儿童出生登记、食品卫生许可等。
信息发布类统计数据集:如中国卫生统计年鉴中卫生机构设置、卫生人员资源分布、卫生经费的筹集及分配等数据集,各类卫生机构的统计月报、年报等。
为满足特定目的收集整理制作的数据集:包括通过调查、监测、检测、实验等方式获取的满足科学研究、业务咨询或卫生服务决策等需求的数据集。如医药卫生数据共享数据集、卫生服务调查数据集、疾病及危险因素调查数据集等。
③数据集标识符编码规则:数据集标识符编码采用字母和数字混合编码,其结构为“DCC.VI”,其中DCC是数据集类目编码,采用长度9位的字母和数字混合编码,按业务领域代码,一级类目代码,二级类目代码,顺序按从左到右排列,结构图如图13-5。
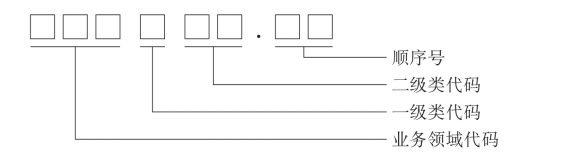
图13-5 数据集标识符编码规则
“VI”是版本标识符,版本标识符由四部分组成:“V”+“m..m”+“.”+“n..n”,其中“m..m”表示版本号,“n..n”表示次版本号,“m..m”和“n..n”由阿拉伯数字组成。如果数据元更新前进可以进行正常的数据交换,则更新后主版本号不变,次版本号等于当前版次号加1。如果数据元更新前后不能进行正常的数据交换,则更新后主版本号等于当前主版本号加1,次版本号置0。
④数据元描述规则:数据集由数据元组成,数据元在特定数据集中的唯一标识代码用内部标识符表示,采用长度13位的字母数字混合码,其中包含2位小数点,内部标识符如图13-6所示。
其中,数据元顺序号用3位阿拉伯数字表示,代表数据元在特定数据集中的序号,从001开始顺序编码,数据元顺序号与数据集分类编码之间用“.”分隔。
(2)电子病历数据标准。2009年,国家卫生部颁布了《电子病历基本架构与数据标准(试行)》,它是我国卫生领域制定、发布的首部国家级具有中西医结合特点的电子病历业务架构基本规范和数据标准。主要包括两部分内容,第一部分是“电子病历基本架构”,第二部分是“电子病历数据标准”,其中,电子病历的数据标准包括四项内容:电子病历数据结构、电子病历临床文档信息模型、电子病历临床文档数据组与数据元标准、电子病历临床文档基础模版与数据集标准。
①电子病历数据结构:电子病历主要由临床文档组成,临床文档中的数据存在着一定的层级结构关系,其中有包含与被包含的关系,也有按同类属性相互嵌套的关系。电子病历数据结构用于规范描述电子病历中数据的层次结构关系,即电子病历从临床文档到数据元的逐步分解,或从数据元到临床文档的逐步聚合关系。电子病历数据结构分为四层(图13-7)。
图13-6 数据元描述规则

图13-7 电子病历结构图
临床文档:位于电子病历数据结构的最顶层,是由特定医疗服务活动产生和记录的患者临床诊疗和指导干预信息的数据集合。如:门急诊病历、病案首页、会诊记录等。
文档段:结构化的临床文档可拆分为若干逻辑上的文档段,为构成该文档段的数据提供临床语境。文档段由数据组组成,并通过数据组获得特定的定义。
数据组:由若干数据元构成,数据元集合体构成临床文档的基本单元,具有临床语义完整性和可重用性特点。
数据元:是可以通过定义、标识、表示和值域等一系列属性进行赋值的最小、不可再细分的数据单元,它位于电子病历数据结构的最底层。
②电子病历临床文档信息模型:临床文档信息模型是为电子病历中不同来源和用途的医疗业务活动记录建立标准化的数据表达模式和信息分类框架,实现临床文档的结构化表达和数据元的科学归档,方便电子病历信息的快速理解和共享。
临床文档由文档头和文档体组成,文档头可理解为临床文档的元数据,用于临床文档交换与共享时的标识、定位和管理。文档体是临床文档的具体记录内容。文档头和文档体分别由承担不同角色和作用的两级嵌套结构数据组构成。其中,文档头由10个一级数据组和2个二级数据组构成;文档体由16个一级数据组和48个二级数据组构成。
③电子病历临床文档数据组与数据元标准
数据组标准:数据组是构成临床文档的基本单元,是按一定的业务规则将相关数据元聚集、形成的一种复合数据结构。依据WS/T 303-2009卫生信息数据元标准化规则,数据组标准规定了电子病历临床文档中所有数据组的分类代码和数据元构成。根据电子病历基本内容规范和临床文档信息模型,共制定了数据组76个。
数据元标准:数据元是电子病历数据标准化的最小单元,分为简单数据元和复合数据元两种类型。数据元的命名以及相关属性定义必须遵循统一的卫生信息数据元标准,才能进行无歧义的信息交换和协调运作。针对电子病历临床文档中的76个数据组,共制定了465个数据元和76个数据元值域代码表。
④电子病历临床文档基础模板与数据集标准
临床文档基础模板:临床文档基础模板是根据临床文档信息模型以及各类医疗服务活动的业务规则,通过对数据组和数据元的基数约束以及数据元允许值约束,生成各类实际应用的结构化、标准化临床文档,保证电子病历数据采集和交换的一致性,它是用于指导临床文档数据创建的形式和方法。
一个临床文档基础模板可生成对应的一个或多个临床文档,其基本构件是可重用的数据组、数据元及数据元值域。针对医疗服务中与电子病历相关的14类、57项业务活动记录,共制定了17个临床文档基础模板。
临床文档基础模板数据集标准:临床文档基础模板数据集标准是对临床文档基础模板中所包含的各数据组和数据元,按照统一的属性描述规则进行有关限制性说明,包括数据元及数据元值域代码标准。一个基础模板一般对应一个数据集,共制定了临床文档基础模板数据集标准17个,其中包括3个住院病程记录基础模板数据集。
(3)健康档案数据标准。2009年,国家卫生部发布了《健康档案基本架构与数据标准(试行)》,主要包括两部分内容。第一部分是“健康档案基本架构”,第二部分是“健康档案数据标准”,其中,数据标准部分又包括健康档案相关卫生服务基本数据集标准、健康档案公用数据元标准、健康档案数据元分类代码标准。健康档案的各项标准是一个不断完善的过程,将随着医疗业务发展和实际需要不断补充和完善。
①健康档案相关卫生服务基本数据集标准:与健康档案相关的每一个卫生服务活动均对应一个基本数据集,基本数据集标准规定了数据集中所有数据元的唯一标识符、名称、定义、数据类型、取值范围、值域代码表等数据元标准,以及数据集名称、唯一标识符、发布方等元数据标准。
该标准已制定了32个健康档案相关卫生服务基本数据集标准。按照业务领域分为3个一级类目:基本信息、公共卫生、医疗服务。其中“公共卫生”包含4个二级类目:儿童保健、妇女保健、疾病控制、疾病管理。
表13-1列出了健康档案相关卫生服务基本数据集标准部分目录,如:《儿童健康体检基本数据集》的数据集标识符为“HRB01.03”,表示该数据集标准属于“健康档案领域(HR)”中的一级类目“公共卫生(B)”下的二级类目“儿童保健(01)”,数据集顺序号为“03”。
②健康档案公用数据元标准:健康档案32个相关卫生服务基本数据集中共包含2252个数据元。其中两个或两个以上数据集中都包含的数据元,称为公用数据元。健康档案公用数据元标准中共包含公用数据元1163个,191个数据元值域代码表。公用数据元是不同业务领域之间进行无歧义信息交换和数据共享的基础。健康档案公用数据元标准规定了健康档案所必须收集记录的公用数据元最小范围及数据元标准,目的是规范和统一健康档案的信息内涵和外延,指导健康档案数据库的规划设计。
表13-1 健康档案部分数据集

③健康档案数据元分类代码标准:从信息学角度对数据元进行科学分类和编码,目的是为健康档案中来源于各种医疗卫生服务记录的所有信息(数据元),建立一个统一的、标准化的信息分类框架,使得不同的信息(数据元)根据其不同的特性,能够分别定位和存储在相应的层级结构中,方便健康档案信息利用者的快速查找和共享。
(二)数据中心建设
1.临床数据中心 随着卫生信息化建设的深入,内部的信息化建设逐步加强,但医疗机构之间的总体协同效果差,没有统一的数据交换标准,缺乏数据共享相互协作的机制,没有统一的数据采集交换平台。医疗机构之间数据普遍存在数据结构不一致、数据不完整、医疗数据不能进行有效共享和交换等一系列问题。研究型医院临床数据中心(Clinical Data Repository,CDR)的主要任务就是获取各个医院各信息系统产生的数据,并进行数据标准化处理,为数据交换和共享做准备。它通过统一的数据标准定义、统一的数据中心构架以及集中的数据管理,实现在众多异构数据库中进行数据采集、数据标准化、数据分析、数据集成和利用,从而达到临床医疗数据共享和交换的目的。
(1)CDR的架构。临床数据中心包括四个层面的建设内容,底层是数据存储层,实现数据的统一存储和管理;中间层是数据服务层,通过数据服务层的数据访问接口实现对数据库的读写操作,这些服务接口作为医疗临床信息的服务总线对外提供访问服务,起到隔离和解耦的作用,保障数据的安全和机密性;顶层是数据展现应用层,通过专业的集成视图直观形象地展现临床数据中心的数据;还有一层是采集层,采集层起到的作用从分散在医院各个角落的信息源采集需要的临床数据到临床数据中心,这些采集接口作为医疗临床信息的数据总线进行管理。整体系统架构图如图13-8所示。
底层的数据存储层包括在线生产库、ODS长期存储库、临床文档库和多媒体医学影像存储库四个组成部分,分别负责不同类型临床数据的存储和管理。
①在线生产库:在线生产库是主要用于支撑日常医疗业务运作,基本包括一体化医护工作站集成终端Pride和所有基础HIS系统,如患者登记、挂号、收费、取药等。随着多年的系统应用,在线生产库的数据规模日渐增大,为了优化性能,通常的做法是将统计周期之外的历史数据迁移出该库,如一年之前的检验结果数据。这样也使得很多在临床医疗过程中,对于一线医护工作者甚具参考价值的历史病历信息无法被实时地获取。为此,建设临床数据中心一个重要的工作就是,建设一个能有效存储和管理这些历史病历信息,即ODS长期存储库。
②ODS长期存储库:ODS长期存储库作为一个长期永久存储和管理历史病历信息的关系型数据库,不仅仅存储上述从在线生产库迁移出的历史数据,也包括在不同时期建设的信息系统,由于系统的更新换代,这些被弃用的信息系统中存储了大量有价值的临床信息。为此,将这些不同时期、不同厂商的信息系统的临床数据,经过必要的清洗、术语转换,最终永久存储到ODS库。根据从生产库到ODS迁移的策略不同,一个患者的所有临床数据根据所属就诊的状态不同,分别分布在生产库和ODS库,两者之间可能会存在一些冗余,但两者之和将覆盖该医院所有临床数据。
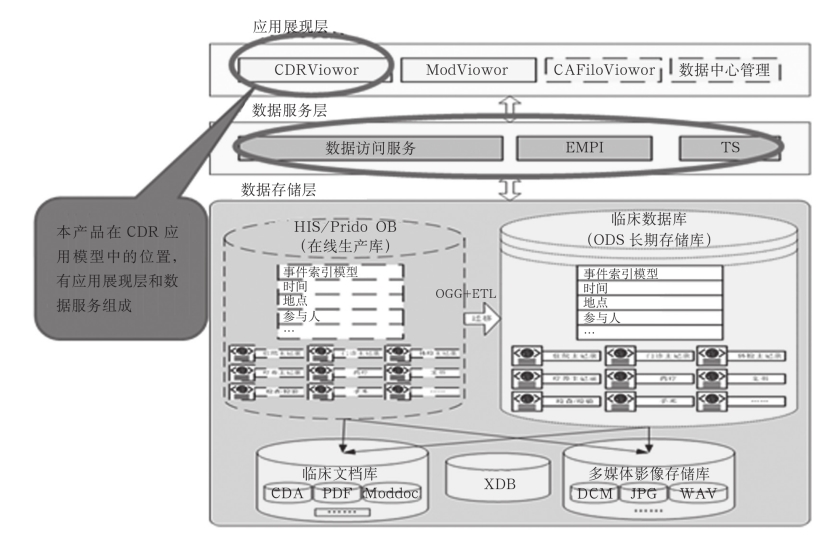
图13-8 临床数据中心架构
③临床文档库:在现阶段,受限于电子病历标准化和结构化技术的发展,病历文书中的大部分内容还无法实现完全结构化,仍然只能以自由文本的形式进行表达,以文件为单位进行存储和管理。这些文档可能包括使用Meddoc文书编辑器产生的临床病历文书;用于跨医疗机构文档交换的符合国际标准的CDA格式文档;以及引入数字签名CA技术后,真正实现无纸化存储的类PDF文档,CDR将使用专门设计的文件服务器存储这些临床文档。
④多媒体医学影像存储库:除了存储于关系型数据库中的结构化数据和文件格式的病历文书以外,医学影像(包括静态和动态)是一类比较特殊的临床数据。通常,医院在建设院级PACS系统时,就建有院级影像中心,这类数据大部分已得到影像中心的统一管理。同时,一方面,它们的数据量非常庞大,在患者的一次就诊过程中就可能会生成数以千计的CT图像,可能还包括各种高分辨率的CR、DR影像,可以预见,随着医学成像设备地不断发展,数据量还会越来越大;另一方面,在医学影像领域,PACS相关的数据存储技术已经非常成熟,DICOM标准也得到了广泛的应用,大量医学影像的应用软件都在不同程度上支持DICOM标准接口。出于这两方面的考虑,在建设CDR时通常不会在物理上重建一个影像中心,因为相关硬件投入是巨大的,CDR中将管理已有影像中心中所有影像的访问索引。而对于那些未纳入已有影像中心管理的多媒体影像文档,如心电检查的波形数据,部分医技辅诊系统产生的动态视频(如内镜检查录像、手术过程录像),这些文档需要在CDR中存储或管理,CDR提供专门设计的文件服务器存储。
(2)临床数据中心数据存储内容。在我国现行的《病历书写基本规范》中,明确了病历是指医务人员在医疗活动过程中形成的文字、符号、图表、影像、切片等资料的总和,其内容包括门(急)诊病历和住院病历。该规范中又进一步说明了门(急)诊病历内容包括门诊病历首页(门诊手册封面)、病历记录、化验单(检验报告)、医学影像检查资料等;住院病历内容包括住院病历首页、住院志、体温单、医嘱单、化验单(检验报告)、医学影像资料、特殊检查(治疗)同意书、手术同意书、麻醉记录单、手术及手术护理记录单、病理资料、护理记录、出院记录(或死亡记录)、病程记录(含抢救记录)、疑难病例讨论记录、会诊意见、上级医师查房记录、死亡病例讨论记录等。临床数据中心中既要存储门诊、住院等临床数据,还要包括体检的健康数据,为此,临床数据中心将针对具体需求,对数据内容进行建模和数据库表的设计。
(3)CDR数据整合。由于医院各个信息源对数据有各自的定义,同时也分散在医院各个角落,为了建立以患者为中心的集中式临床数据中心,因此不可避免涉及数据的整合利用。
①数据同步(Oracle GoldenGate):在线生产库和ODS(Operational Data Store,ODS)长期存储库之间数据同步策略按时效性可以分为两种,实时同步和延时同步。实时同步就是HIS和CIS等业务系统基于在线生产库的任何业务操作所产生的医疗数据都将在毫秒级的时间内同步到ODS长期存储库;延时同步方式,两库之间数据同步的时机可能是在患者就诊完毕或每天某时刻(如深夜)定时同步。目前为了能及时展现患者的诊疗信息,这里我们推荐采用实时同步方式。鉴于同步内容数据量大,实时性要求高,这种应用场景下已不适宜采用基于集成平台的通过消息交互的应用集成方式。消息集成方式往往需要一个发起方和接受方,而发起方和接受方往往需要一些额外的支持,如发起方需要调用接受方提供的接口等,期间可能还涉及一些消息转换、反馈、确认等复杂的来回交互,最主要的是,消息集成在数据量很大的情况下,处理速度不是很快,因此,我们这里将使用数据集成的方式来实现数据同步。
为了避免抽取过程对生产库造成不必要的性能影响,这里会结合同步工具(Oracle GoldenGate)使抽取过程对生成库的影响降低到最小,采用Oracle GoldenGate同步的最大的好处是几乎不会增加对生产源的任何负载,同时还很灵活的决定从什么时候开始抽取同步、还可以重复抽取和分发。
Oracle GoldenGate涉及需要数据同步的包括两个部分:在线生产库和ODS长期存储库映射区。我们采用GoldenGate实现在线生产库和ODS长期存储库之间的数据同步。其基本结构图如图13-9所示。
从上图可以看到发生在在线生产库上的相关数据变化通过GoldenGate实时同步到ODS长期存储库。Oracle GoldenGate将在线生产库变化数据捕获、传递和复制到ODS长期存储库。当HIS数据库发生数据变化的时候,如医嘱下达、校对医嘱之后,此时运行在HIS数据库服务器上的GoldenGate将捕获该功能业务对应的变化数据,并通过网络传递到ODS长期存储库,ODS数据库接收到这些变化数据之后,运行在ODS数据库服务器上的GoldenGate解析这些变化数据并应用到ODS数据库。
通过GoldenGate可以很好地实现在线生产库和ODS长期存储库之间的独立和联系,使他们各尽其职,分工明确,一起很好地共同支撑整个医院的正常运营。

图13-9 数据同步
②ETL(Extract Transform Loading,数据抽取转换装载):数据整合意味着需要对数据进行抽取、转换和加载等工作,这其实是完整的ETL过程。ETL负责将分布的、异构数据源中的数据如关系数据、平面数据文件等抽取到临时中间层后进行清洗、转换、集成,最后加载到ODS中,成为联机分析处理、数据挖掘的基础。该工作是CDR实施过程中最重要也是最烦琐复杂的工作,占到整个CDR设计工作的60%~80%作用量,甚至更多。
数据整合的基本过程:首先使用Oracle GoldenGate将生成库的数据变化实时同步到ODS库的数据映射区域,然后再对变化的数据进行ETL处理,最终将数据按照特定的规则和要求装载到临床数据中心。
经过对ETL工具成本、易用等角度的考察,这里我们会采用Kettle进行数据抽取。Kettle是一款国外开源的ETL工具,纯JAVA编写,绿色无需安装,数据抽取高效稳定。Kettle中有两种脚本文件,Transformation和Job,Transformation完成针对数据的基础转换,Job则完成整个工作流的控制。
(4)基于临床数据中心的展示应用。在临床数据中心的基础上,通过开放式的医疗信息平台以及电子病历集成终端,为医护人员提供一个更方便、快捷、准确和智能化的数字工作环境。
①电子病历集成视图(CDRViewer):电子病历集成视图意在通过一个图形化的界面,即可方便、快捷、准确地将患者的病历资料、各种影像资料、检查、检验资料集中展现,实现快速浏览,极大地提高工作效率。系统如图13-10所示。
电子病历集成视图为医、护操作者在一个界面上,提供了可查询、浏览各医护文书、特护记录、辅诊检查资料的快捷方式。且界面以直观方式显示患者当前各生命体征(体温、脉搏、血压、呼吸)、检查检验、医嘱等患者重要的观察指标,并能以时间方式查询此前任意上述指标的情况、相互关系和趋势。在该集成视图中,各种电子病历数据的前后、因果关系一目了然,医护人员不仅可以观察患者的上述各类指标,从整体上把握其病情发展情况,还可以直观地查阅在病情不断变化的情况下,对患者所进行的各种处置护理情况,诊疗计划的制定、执行情况及其临床效果等等,同时也可以轻松地翻阅患者的历史病历数据,为下一阶段的诊疗工作提供极其丰富的参考信息。这样的集成视图真正体现了“以患者为中心”的观点,在很大程度上改善了传统形式病历固有的缺陷与不足。
②数据综合浏览视图:数据综合浏览视图用于实现对各种医学影像(X线、CT、MRI、超声、胃肠镜)、心电图、监护数据和麻醉监护数据等在内的多种医疗数据的综合阅览分析,在大量富有价值的参考信息的基础上,开展下一步的诊疗工作。该视图完全打破了科室的界限,使全院所有医生、护士以及医务人员均采用统一的界面进行多种医疗数据的浏览和操作,改变了各科室数据分散而孤立的现状,跨科室实现了所有医疗数据的集成和整合。借助于数据综合浏览视图,医护人员可以对集中视图中任意部分进行独立显示。系统如图13-11所示。

图13-10 时间轴连续临床数据集成视图
2.影像数据中心 随着数字化医学影像设备在医学诊断中的应用越来越广泛,它与临床各学科的结合也日益紧密,在医疗诊治工作中发挥着相当重要的作用,而基层医疗机构医学影像设备缺乏和诊断能力相对较低的现状也一直存在。许多医院在建成了全院PACS系统后,医学影像数据呈海量增长,其管理和使用成为急需解决的问题,因此,以研究型医院建设为契机,构建影像数据中心,是解决这一难题的有效途径。
影像数据中心建设的总体目标是实现跨部门、跨机构、跨区域的数字医学影像的数据存储与广泛共享。通过医学影像数据中心数据区域化共享和利用,各个医院之间可以互相调阅被作为重要诊治依据的医学影像资料、诊断结果、电子病历等相关医疗数据,对患者在不同医院和不同时期的就诊信息进行跟踪和比对,有利于避免重复检查,降低医疗成本,提高治疗效果。汇集到医学影像数据中心的数据还可以为更多的机构服务,如医学院可调阅数据中心医学影像数据,起到了医学影像教育和培训平台的功能。科研人员可以根据需要调阅影像数据中心某类疾病的影像资料,对该疾病的发展演变研究提供影像数据支撑。
(1)影像数据存储方式。医学影像数据中心的建立解决了医学图像数据存储、管理和共享的难题,在集中统一存储和管理的架构下,依据影像数据存储要求和数据量,采用三级存储结构,即影像在线存储、近线存储、离线存储三级,三级存储方式在一定程度上很好地解决了海量影像数据的长期存储与管理问题。
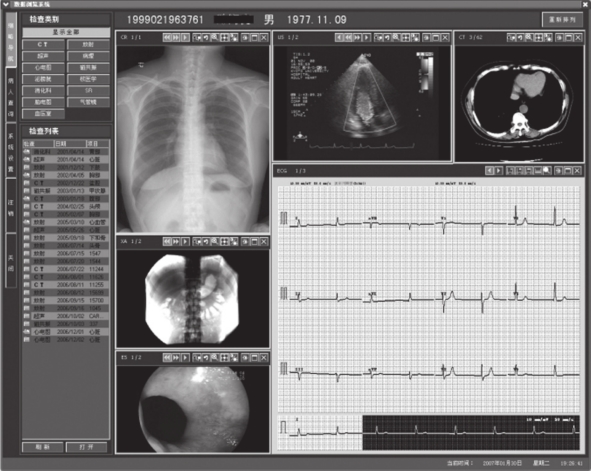
图13-11 数据综合浏览视图
①在线存储:用于集中存储区域内各医疗单位近期内(如3个月)的在线影像数据,供各医院医生、科研和教育工作者等快速方便地通过网络实现医学影像数据的调阅。在线存储需要大容量、高性能存储器,能满足用户高频率的访问、读取、写入请求。
②近线存储:某一时刻存储设备中只有少数影像数据在线存储供用户随时访问,对于各医疗机构产生的超过一定期限、用户访问频率相对较少的影像数据采用近线存储方式,在用户需要访问采用近线存储的影像数据时,通过各种优化算法将其加载到系统中,实现安全、及时、准确的数据访问。近线存储的影像数据精确定位及存取所耗费的时间较长,需要采取一定的手工操作和管理。
③离线存储:对于医疗机构产生的时间较长(5年以上)的过时医学影像数据交由医疗影像数据中心统一存储和管理。医院的PACS系统与医学影像数据中心管理平台相连接,通过VPN专线将影像数据定时上传至中心。离线存储在安全性、大容量等方面的要求更高,存储介质可采用磁带库和光盘,将影像数据刻录在磁带或光盘存储介质上,按时间顺序保存,实现医学影像资料的永久性海量存储与管理。
(2)影像和数据访问解决方案
①数据流分析:当患者在放射科进行登记后,影像设备即可通过访问所属前置服务器来取得RIS中存储的患者Worklist信息,并将检查状态反馈给前置服务器,前置服务器随之更改保存在中心服务器RIS系统中的患者检查状态。
当患者完成检查,技师将图像发送给设备所属前置服务器后,前置服务器对图像数目进行确认,并与RIS中保存的患者文本数据进行匹配,确保患者信息与图像信息的一致性;随后前置服务器对图像进行标记,并将图像压入图像归档队列;安装于各个前置服务器上的队列管理器实时监测图像归档队列,按照预定义规则将队列中的图像送至中心服务器,并对归档过程进行监控,当发现归档失败时队列管理器可以自动按照预定义规则处理错误,以保证中心服务器和前置服务器之间数据的一致性;归档过程完成后,在医生调阅患者本次或历史影像时,所有的调阅请求均先提交给中心服务器调度管理程序,调度管理程序查阅中心数据库中保存的数据迁移记录,查询图像是否已从前置服务器迁出或从中心服务器迁回,按照就近分配原则指示客户端从前置服务器或中心服务器上取得所需图像,通常情况下的图像分配策略是,患者本次检查的各种图像(一次检查可以包括不同类型,如既做CT又做超声或MRI)将从所做检查对应科室前置服务器上获得,患者历次检查的所有图像将从中心服务器(无预约时)或所属科室前置服务器上获得(患者提前预约时)。
②数据一致性的保障措施:服务器集群系统实际上采用了一种集中+分级的混合型结构,综合了分布式系统网络负载均衡和集中式系统数据统一管理的优点,同时避免了分布式系统数据不一致和集中式系统所有终端直联服务器导致网络和中心节点负载过大的问题。
在服务器分级体系架构中,可以通过以下两个策略保证节点之间数据的一致性。
首先,所有用户可以访问的数据(报告和图像)索引只保留一份,均直接存储在中心服务器上,客户端直接与中心服务器进行交互,然后按照中心服务器的指示从集群内指定节点上(通常是前置服务器)取得所需数据,这样既可以保证不同前置服务器所属的终端客户均访问的是同一份数据,又可以保证因图像传输而产生的大量数据流均保持在科室服务器和所属工作站之间,大大节省了中心网络带宽。
其次,所有可能导致在多个节点之间需要进行同步数据的更新操作(如管理员手动修改了图像中的姓名,医生将对图像所作标注保存到服务器上等)也直接由客户端向中心服务器提交,中心服务器收到数据更新请求后,首先按照操作内容更新本地影像数据,并修改中心数据库中相应数据记录,以保证所有对已修改数据的访问请求均直接从中心服务器本地影像中获得响应,随后再将所作更新操作广播到集群中每个节点上,保证整个集群内部数据的一致性;即使因为网络故障等原因导致个别节点的更新操作不能进行,系统也可以日志方式通知管理员,同时由于更新操作涉及的图像均可以直接从中心服务器本地阵列中获得,该部分“脏”数据用户永远也不可能访问到,从而确保了整个系统数据的统一。
(三)数据专库建设
1.慢性病随访库 慢病随访是通过专业的技术手段定期向慢病患者发送与其疾病相关的随访内容,掌握其疾病康复和治疗情况,并在用药、饮食、运动等各方面进行专业性的指导,帮助患者快速、有效地进行疾病控制和治疗。研究型医院可以建立慢病随访库,采集来自不同医疗机构或卫生管理部门上传的慢病数据,患者本人也可以自己采集体征数据并上传到慢病随访库。通过建立慢病随访库,可以对采集的慢病数据进行科学管理和利用,为预测慢性病发展趋势、评价治疗和防控效果提供科学依据,为医院制定慢性病控制决策提供监测指标。
慢病随访库的数据可以通过医院HIS系统或社区卫生服务平台将数据上传到重点慢病随访库;没有医院HIS系统、社区卫生服务平台的地区可以由录入人员手工录入慢病收集的信息到系统中,慢病患者自己也可以在家里通过穿戴式设备、远程心电监护仪等设备将血压、心跳、血糖等生理参数通过网络发送到慢病随访库。用户可以根据权限对慢病随访库的数据进行查询、查看、修改、删除、合并等操作,并可根据慢病患者的情况进行随访信息的录入。
慢病随访库数据资源体系包括如下内容。
(1)基本数据集:①脑卒中、恶性肿瘤、高血压、糖尿病、冠心病等重点慢病个案最小数据集,包括人口学信息和疾病相关信息等;②审核、订正基本数据集;③质控数据集,包括及时率、完整率等。
(2)监测指标数据:包含时间、空间、人群分布维度的以下监测指标。①发病率:指选择的报告日期范围内的发病数/相应地区当年平均人口数×100%。②患病率:指患病数/相应地区当年平均人口数×100%。③慢病死亡率:指选择的死亡日期范围内的死亡数/相应地区当年平均人口数×100%。
(3)管理指标数据:包含时间、空间、机构分布维度的以下监测指标。①管理率:指管理数/相应地区(高血压或糖尿病等)的患者的人数×100。②规范管理率:指规范管理数/相应地区(高血压或糖尿病)的患者已管理的人数(已管理指患者有随访信息)×100。③控制率:指控制数/相应地区(高血压或糖尿病等)的已管理的患者人数(已管理指患者有随访信息)×100。
2.生物标本库 近年来大量生物学实验的数据积累,形成了当前数以百计的生物信息数据库。它们各自按一定的目标收集和整理生物学实验数据,并提供相关的数据查询、数据处理的服务。随着因特网的普及,这些数据库大多可以通过网络来访问,或者通过网络下载。
一般而言,这些生物信息数据库可以分为一级数据库和二级数据库。一级数据库的数据都直接来源于实验获得的原始数据,只经过简单的归类整理和注释;二级数据库是在一级数据库、实验数据和理论分析的基础上针对特定目标衍生而来,是对生物学知识和信息的进一步整理。国际上著名的一级核酸数据库有GenBank数据库、EMBL核酸库和DDBJ 库等;蛋白质序列数据库有 SWISS-PROT、PIR 等;蛋白质结构库有PDB等。国际上二级生物学数据库非常多,它们因针对不同的研究内容和需要而各具特色。下面简要介绍著名的基因和基因组数据库。
(1)GenBank 数据库。GenBank数据库包含了所有已知的核酸序列和蛋白质序列,以及与它们相关的文献著作和生物学注释。它是由美国国立生物技术信息中心(NCBI)建立和维护的。它的数据直接来源于测序工作者提交的序列;由测序中心提交的大量EST序列和其他测序数据;以及与其他数据机构协作交换数据而来。GenBank每天都会与欧洲分子生物学实验室(EMBL)的数据库、日本的DNA数据库(DDBJ)交换数据,使这三个数据库的数据同步。GenBank的数据可以从NCBI的FTP服务器上免费下载完整的库,或下载积累的新数据。NCBI还提供广泛的数据查询、序列相似性搜索以及其他分析服务,用户可以从NCBI的主页上找到这些服务。
GenBank库里的数据按来源于约160000个物种,其中约17%是人类的基因组序列(所有序列中的64%是EST序列)。每条GenBank数据记录包含了对序列的简要描述,它的科学命名,物种分类名称,参考文献,序列特征表,以及序列本身。序列特征表里包含对序列生物学特征注释如:编码区、转录单元、重复区域、突变位点或修饰位点等。所有数据记录被划分在若干个文件里,如细菌类、病毒类、灵长类、啮齿类,以及EST数据、基因组测序数据、大规模基因组序列数据等18类,其中 EST 数据等又被各自分成若干个文件。
GenBank flatfile(GBFF)是GenBank数据库的基本信息单位,也是最广泛地用以表示生物序列的格式之一。DDBJ flatfile格式与GBFF格式是相同的,EMBL格式则与之有所差异。所有这些格式实际上都是由更结构化的ASN.1生成的。但是主要由于历史的原因,许多用户在工作中使用GBFF。
GBFF可以分成三个部分,头部包含关于整个记录的信息(描述符),第二部分包含了注释这一记录的特性,第三部分是核苷酸序列自身。所有的核苷酸数据库记录(DDBJ/EMBL/GenBank)都在最后一行以“//”结尾。
头部是记录中与数据库关联最大的部分。各个的数据库并不一定在这一部分包含相同的信息,而可能存在着微小的差别。但各数据库已作出努力以在彼此之间保证信息兼容。所有的GenBank flatfile开始于LOCUS行。这一行中的第一项是LOCUS名称。历史上曾用这个名称来表示本记录描述的基因座,提交者和数据库工作人员花费了无数的时间来设计这一名称。这一成分开始于一个英文字母,总长度不能超过10个字符。第二个字符以后可以是数字或字母,所有字符均要大写。LOCUS名称在以前是最为有用的,那时大多数DNA序列记录只表示一个基因座,这样在GenBank中寻找一个可以用少数几个字母和数字来代表生物体的独特的名字是很容易的事。为了可用起见,LOCUS名称在数据库中必须是独一的。因为几乎所有有意义的命名符都被使用过了,所以今天LOCUS名称已不再是一个有用的成分。LOCUS行中的下一项表明生物分子的类型。“分子类型”通常是DNA或RNA,但也有少量其他类型出现,以表明生物分子的最初来源。
LOCUS行中的日期是数据最后被公开的日期。在许多情况下,也是第一次被公开的日期。记录中包含的另一个日期是序列提交给数据库的日期。
DEFINITION行(也称为“DEF”行)在GenBank记录中用以总结记录的生物意义。这一行将出现在NCBI的FASTA文件中,这样任何人进行BLAST相似性搜索时都会看到这些信息。但是,用一行文字来说明生物背景并不总是可行的,对此不同的数据库采用了各自的解决方法。其中有一些共识,并且每个数据库也都了解其他数据库的解决方法,并尽力与之一致。
检索号在记录的第三行,是从数据库中检索一个记录的主要关键词。这个号码将在参考文献中被引用,并始终和序列在一起。就是说,当序列被更新(例如更正一个核苷酸)时,这个号码不会改变。检索号码采取下列两种方式之一:1+5或2+6格式。1+5格式是指1个大写字母后跟5位数字;2+6格式是指2个大写字母后跟6位数字。绝大多数新近加入数据库的记录采取后一种方式。所有的GenBank记录都只有一个单独的ACCESSION行,行中可能有多个检索号码,但绝大多数情况只有一个检索号。这通常称为主检索号码,其余的是二级检索号码。例如:AF010325.1,这表明序列第1版,检索号为AF010325,gi号为2245686。
KEYWORDS是另一个有趣的历史遗留物,并且不幸地在很多情况下被误用了。给一个记录加上关键词通常并不十分有效,因为在过去的年月中有许多作者选用了不在受控词表中的词,并且在整个数据库中用法也不一致。因此,NCBI不鼓励使用关键词,但在查询时加入关键词是可以的,特别是那些没有在其他记录中出现过的词,或以一种受控的方式来使用的词(例如:对于EST,STS,GSS,HTG记录)。
SOURCE行中有生物的通用名或科学名称。有些情况下也有其他来源的信息。现在正在一致努力以保证来源特性中包含所有必须的信息(不同于现在的SOURCE行),并且所有关于分类的信息(SOURCE行和ORGANISMS行)可以从来源特性以及NCBI分类服务器中获得。对于系统族或关于分类的其他方面感兴趣的读者可以访问NCBI的分类主页。这一分类被所有核苷酸序列数据库以及蛋白质数据库Swiss-Prot所采用。
每个GenBank记录至少要有一篇参考文献,许多情况下有多篇。未发表的论文标记为“未发表”或是“已投”,如果将来文章发表的话则将代替于此。参考文献提供了科学证据以及一个背景来解释这个特定的序列为何会这样确定。当参考文献发表时,通常会有一个MEDLINE标识符,正如下面例子中一样,提供了指向MEDLINE/PubMed数据库的链接。在1998年末,又加入了一个新的行,以及其标识符PubMed,允许指向PubMed数据库以及发表者在线全文电子版的链接。
GBFF记录的中间部分,也是最重要的一部分,就是注释,它直接表达了记录的生物背景知识。也许有人争辩说生物背景在记录所引用的参考文献中有最好的表现,但不论怎样,记录中的一整套注释有助于快速地抽取相关生物信息,并允许提交者指出这一记录当时为什么会被提交到这个数据库中。这里对于注释的选择就十分关键了。特性表文档详细描述了合法的特性(允许使用的注释),以及这些特性的允许限制词。不幸的是,这里经常有一些非法的,推测性的或由计算得出的注释。如果一个注释仅是由计算得到的,它作为记录说明的可用性就大打折扣了。
来源(source)是唯一一个必须在所有GenBank记录中出现的特性。所有的特性都有一系列合法的限定词,有些是强制性的[例如来源中的/organism(生物体)]。所有的DNA序列记录都有出处,即使是合成序列这样极端的特例也一样。大多数情况下一个记录只能有一个来源特性,并带有/organism限定词。限定词organism包含属和种的科学名称,有些情况下还可以在亚种水平描述。
CDS指示读者如何将两个序列连接在一起,或如何根据核苷酸序列以及基因编码得到氨基酸序列。GBFF以DNA为核心,通过DNA序列坐标系统映射所有特性,而不是从氨基酸的角度。在分析这些数据时,我们必须从DNA坐标推导出氨基酸位置,并且我们对于所编码蛋白质的了解也将仅限于从对DNA特性的描述中获得。这一限制可被Sequin克服。这一例子也显示了数据库交叉索引(db_xref)的使用。这一受控限制词允许数据库将另一个外部数据库的序列(第一个标识符)与一个在本数据库中使用的标识符交叉索引。允许db_xref的数据库都是合作数据库所维护的。正如上面提到的,NCBI给每个记录赋予一个gi(geninfo)标识符。这意味着翻译产物蛋白质序列(不是简单附属于DNA记录,如同在GenBank记录中显示的),也有自己的gi号码。一个特定的标识符当且仅当序列更改时才更改。蛋白质gi号码现在作为PID db_xref或蛋白质标识符出现。
(2)GenBank二级数据库
①表达序列标记数据库dbEST:EST(Expressed Sequence Tags)方法已被证明是识别转录序列的最有效方法。在1990以前,关于人类基因序列的数据主要来自于对单个基因的研究,EST数据的出现是生物信息学发展历史上的一个里程碑。EST序列大约覆盖了人类基因的90%。EST序列中含有大量的基因信息,利用这些信息可以发现新的基因,阐明基因的功能。dbEST是GenBank的一个部分,该数据库包括不同生物的EST序列数据及其他相关信息,主要是从大量不同组织和器官得到的短mRNA片段。通过WEB页面可以查询有关EST的数据和相关报道,也可以通过FTP下载dbEST数据库。EST数据库的主要作用是通过搜索比较,给实验新得到的一条cDNA序列或基因组序列赋予公认的功能。通过对EST数据库的逆向分析,能识别与疾病相联系的基因。
②基因聚类数据库UniGene:UniGene数据库将GenBank中的序列进行自动分类,形成面向基因群的非冗余集合。每个UniGene群包含代表一个唯一基因的多个序列,附有该基因相关的信息,如基因表达的组织类型、定位图谱。除了基因的序列之外,还包括大量的EST序列。UniGene既可以作为发现新基因的数据源,也可以作为生物学研究人员进行大规模表达分析的辅助工具。需要指出的是,自动分类的过程还有待于进一步发展和完善。目前,UniGene中包括人类、小鼠、水稻、小麦等生物的相关数据,因为这些生物有大量的EST数据。
序列标记位点数据库dbSTS,UniSTS:STS(Sequence Tagged Site)是序列标记位点。dbSTS是NCBI的一个数据源,也是GenBank的一个部分,包含已知的序列标记位点组成和定位信息。可以通过BLAST搜索STS序列,或者直接通过FTP下载序列。
基因组数据库:随着核酸测序技术的迅速发展,人类已经得到一部分生物的全基因组数据,如人、小鼠、大鼠等。这些数据对于我们认识基因组信息组织的奥秘、了解生物体的生长发育的规律是非常重要的。国际上有专门的组织收集和管理这些数据。NCBI基因组数据库Entrez Gonomes所收集的基因组数据量非常大。该数据库还提供了一个基因组数据浏览工具Map Viewer,利用这个工具,用户可以很方便地得到所需要的数据。
单碱基多态性数据库dbSNP:遗传学研究的一个重要方面是建立生物分子序列变化与可遗传表型之间的联系,其中最常见的序列变化就是单核苷酸多态性SNPs(Single nucleotide polymorphisms)。在人类基因组中,在500到1000碱基长度范围内,就会出现一次单碱基的变化。SNPs对人类遗传学研究和医学应用具有重要的意义,无论对于人类种群遗传学的研究,还是疾病易感性分析、药物基因组研究或个体化医疗,都需要深入地研究SNPs。找出人类基因组中所有的SNPs是基因组研究的一个组成部分。某些特定的SNPs等位基因被认为是人类遗传疾病的致病因子,在个体中筛选这类等位基因可以检查其对疾病的遗传易感性。SNPs也可以作为遗传作图的遗传标记,帮助定位和鉴定功能基因。目前,科学家在SNPs筛选和发现方面正在做大量的工作,由于大规模基因组序列分析及其相关技术(特别是基因芯片技术)的不断提高,同时,也由于生物信息学及计算机技术的发展,使得检测和分析SNPs成为可能。
单核苷酸多态性数据库dbSNP是由NCBI与人类基因组研究所合作建立的,它是关于单碱基替换以及短插入、删除多态性的资源库。
(3)EMBL核酸序列数据库。EMBL核酸序列数据库由欧洲生物信息学研究所(EBI)维护的核酸序列数据构成,由于与GenBank和DDBJ的数据合作交换,它也是一个全面的核酸序列数据库。该数据库由Oracal数据库系统管理维护,查询检索可以通过因特网上的序列提取系统(SRS)服务完成。向EMBL核酸序列数据库提交序列可以通过基于Web的WEBIN工具。
(4)DDBJ数据库。日本DNA数据库(DDBJ)也是一个全面的核酸序列数据库,与GenBank和EMBL核酸库合作交换数据。可以使用其主页上提供的SRS工具进行数据检索和序列分析。可以用Sequin软件向该数据库提交序列。
(四)数据化建设对教学和科研的支撑
1.课程数据化 自2012年以来,大型开放式网络课程(massive open online courses,MOOC)日益受到世人的瞩目,它突破了传统课程时间、空间的限制,世界各地的学习者都可以通过互联网在家学习国内外著名高校课程,受到了广泛的欢迎。目前,世界顶尖大学都陆续设立了网络学习平台,在网上提供免费课程。MOOC改变了传统学校传授知识的模式,提供了一种全新的知识传播和学习方式,在教育观念、教育体制、教学方式等方面都有着深刻影响。MOOC大规模、开放和在线的特点,为自主学习者提供了方便灵活的学习机会和广阔的空间。目前比较主流的MOOC平台包括Sakai、Coursera、Udacity、edX等,语言以英语为主。其中,Sakai架构于J2EE之上,具有严格的分层结构和门户系统的功能,支持Plugin机制,它所有的工具都可以看作是Sakai的一个Plugin,因此Sakai不管是应用还是架构上都有着独到的优势。
(1)Sakai应用概述。Sakai是由美国印第安纳大学、密西根大学、斯坦福大学和麻省理工学院于2004年共同发起的一项开源课程管理系统(CMS)计划。该项目的主要目标是,开发Sakai程序的系统架构,将之与已有各种CMS工具和组件整合为一体。这些工具与组件既可用于课程管理,也可作为原有CMS模型的扩展插件,因此与现有的其他CMS产品相比,Sakai更具竞争力。
在中国采用Sakai的案例中使用规模比较大的学校有复旦大学、重庆大学、上海交通大学、第三军医大学、北京邮电大学等。第三军医大学西南医院将开展MOOC课程教育视为研究型医院数据化建设成果应用的重要方面,该院从2013年就采用Sakai作为远程网络教育的教学平台,并承担了西藏等边远地区部队的临床远程教学任务。截止到2014年3月,已经累计有数千人在Sakai平台上进行学习和交流,受到了广大学员的欢迎。西南医院Sakai网络课程充分利用了该院数据化过程中建立的各类医疗数据库、各类专库等数据资源,这些数据资源为开展大型开放式网络课程提供了宝贵的医疗数据来源和数据支撑。
Sakai作为一个帮助师生和研究者创建网络课程学习站点的开源软件,在优化教学上提供了很多方便的工具,不需要懂得HTML知识就可用Sakai提供的工具创建满足自己需要的学习站点,管理自己的学习或教学任务,发布通告信息,开展网络讨论、测试等。

图13-12 Sakai的J2EE框架
(2)Sakai基本架构。Sakai是轻量级的J2EE Servlet容器(container),具有严格的分层结构,即面向用户界面(GUI)的工具集、面向设计人员的API服务/组件,以及面向服务和设计人员并提供工具集、服务和权限的框架(图13-12)。系统采用面向用户界面的工具集与数据层隔离,支持插件机制,每一个工具都可以看作是Sakai的一个插件,通过Spring Beans集合统一接口,提供工具和服务注册的框架以提供各种服务。Sakai服务共分为3种:核心服务、标准服务和接口。其中用于应用开发的Sakai核心服务(Coreservice)有处理用户信息查询(UserDireetoryService)、处理用户会话 (SessionManager)、用户权限检查验证(SecurityService)、站点整合(SiteService)、查找用户位置和工具信息(ToolManager)、为开发新功能和新工具提供注册管理(FunctionManager)等;标准服务有把添加用户信息(UserDirectoryProvider)、增加群用户(GroupProvider)、添加课程和部门相关信息(CourseManagementProvider)等;以控制实体的URL、属性、导入和导出的特殊整合功能接口有添加用户应用实体支持器(EntityBroker/Provider)、创建或者探测事件(EntityBroker/Provider)等。
在插件管理分工中,Sakai基金会负责维护核心插件集合,其余插件作为贡献(Contribution)发布。插件文件结构主要有4个目录,分别是基于接口变量的API、基于安装应用的Impl、基于组件定义的Pack和实现插件界面功能的Too1。
组件是对Sakai服务接口函数的具体实现,Sakai组件管理容器Component Manager借用Spring容器对bean管理来创建、注册和维护Sakai组件,以及对组件生命周期进行管理。为保持各部分自身的独立性和减少对其他层的依赖,禁止不相邻层的直接通讯,Sakai框架采用三层应用架构,它们分别是表现层、业务逻辑层和数据访问层(图13-13)。表现层主要是用户交互、用户界面(GUI)或者是基于浏览器客户端的外观层;业务逻辑层包含信息、数据处理的逻辑规则,也叫中间层;数据访问层主要对数据持久化进行物理存储以及对数据库或者文件系统的访问进行管理。

图13-13 Sakai三层架构
2.科研资源数据化
(1)文献资源数据化。文献资源是指以文字、图形、符号、声频、视频等方式记录在各种载体上的知识和信息资源。它包括:图书、连续出版物(期刊、报纸等)、小册子以及学位论文、专利、标准、会议录、政府出版物等。
文献资源的演变:文献资源信息记录着无数有用的事实、数据、理论、方法、假说、经验和教训,是人类进行跨时空交流,认识和改造世界的基本工具。这类信息经过加工、整理,较为系统、准确、可靠,便于保存与利用,但也存在信息相对滞后、部分信息尚待证实的情况。从整体上说,这类信息是当前数量最大、利用率最高的信息资源。按照各种标准,可以划分出文献的各种类型。按加工情况分,可有一次文献、二次文献和三次文献;按载体形式分,可有书写文献、印刷文献、缩微文献、音像文献、机读文献等;按内容的学科范围分,则有社科文献、科技文献等。一种具体的文献可能具有两种或两种以上文献类型的特征,如《南京大学学报(社科版)》既是期刊,又是一次文献,也是印刷文献和社科文献。国家标准《文献类型与文献载体代码》(GB3469-83)根据实用标准,将文献分成26个类型,即:专著、报纸、期刊、会议录、汇编、学位论文、科技报告、技术标准、专利文献、产品样本、中译本、手稿、参考工具、检索工具、档案、图表、古籍、乐谱、缩微胶卷、缩微平片、录音带、唱片、录像带、电影片、幻灯片、其他(盲文等)。按文献表现形式特征有11类:图书、报刊、学位论文、会议资料、专利文献、科技报告、产品资料、科技档案、标准资料、政府出版物和网络文本。
文献资源数据化建设(document resources information building)是指依据文献信息服务机构的服务任务与服务对象以及整个社会的文献情报需求,系统地规划、选择、收集、组织管理文献资源,建立具有特定功能的文献资源信息化体系的全过程。它是一定范围内的图书馆及其他文献情报机构对文献资源进行有计划的积累和合理布局,以满足、保障社会发展和国家建设的需要的全部活动。
(2)临床资源数据化。生物组织样本库的定义主要指标准化收集、处理、储存与应用健康和疾病生物体的生物大分子、细胞、组织和器官等样本或经处理过的生物样本(DNA、RNA、蛋白等)以及与这些生物样本相关的临床、病理、治疗、随访、知情同意等资料及其质量控制、信息管理与应用系统。通过生物样本库这一重要平台,将基因组学、蛋白质组学和代谢组学等领域大量的基础研究成果快速应用于临床,真正实现转化医学的目标:理论与实际相结合;基础与临床相整合;实现分子、细胞、结构、功能、表型、发病机理、生理病理、环境遗传、预警诊断、预防治疗、医学信息的系统分析,并最终提高临床诊疗水平。对恶性肿瘤等重大疾病的研究起着至关重要的推动作用,是众多重要科研成果快速产业化、应用到临床的重要保证。
生物样本库的类型:生物样本库(Biobank)有多种类型,常见的组织、器官库(Tissue bank),如血液库、眼角膜库、骨髓库,到拥有正常细胞、遗传突变细胞、肿瘤细胞和杂交瘤细胞株(系)的细胞株(系)库,近年来出现了脐血干细胞库、胚胎干细胞库等各种干细胞库以及各种人种和疾病的基因组库(Genome bank)。
生物样本库的发展趋势:近年来,美国、欧洲以及国际卫生组织都投入了大量资金建立大型生物样本库。①英国样本库(UK Biobank)。是世界规模最大的人类遗传队列研究样本库。由英国卫生部、医学研究理事会、苏格兰新政院、惠康信托医疗慈善基金会出资启动资金6200万英镑进行建设。全英有20多所著名大学参与建设。已收集样本量50多万份。收集范围包括癌症、心脏病、脑卒中、糖尿病、老年痴呆症等高风险人群。②美国国家癌症中心生物资源和生物样本研究办公室(OBBR)。该组织2005年成立,隶属于美国国立癌症研究所(NCI)。主要作用在于协调内部及外部开展癌症研究项目中有关样本库建设的标准、政策、法规等。③欧洲生物样本库与分子生物资源研究机构(BBMRI)。该机构是由欧洲54个会员机构组成,会员来自欧洲30多个国家的生物样本库相关组织;收集的样本已超过10万份。我国于2003年启动了国家自然科技资源共享平台建设项目,建成一系列具有代表性的生物组织样本库,包括:中国医学科学研究院建立的“中华民族永生细胞库”;国家科技部牵头建立的“中国人类遗传资源平台”;中国医学科学院的癌症组织样本库;国家“重点新药创新”专项临床标本资源库和北京市科委牵头建立的“北京重大疾病临床数据和样本资源库”等。
研究型医院生物样本库建设:从研究型医院的科研管理层面以及科研项目整体规划的角度,探索适合我国国情的研究型医院样本库应用管理体系,并向国内大型综合性三甲医院进行试点推广。研究型医院生物样本库建设内容包括:①基于样本库的科研整体规划和项目流程管理;②临床科室、样本库、科研平台的协调配合管理;③样本使用的评估和监督管理;④配套临床资料、随访信息和研究成果的共享与管理;⑤使用效益的后期监督与评估。在此基础上,制订研究型医院生物组织样本库应用管理规范,开发相关软件,并作为功能模块整合进入已有的医院综合信息管理/生物组织样本库管理系统中,举办应用管理培训班,以期在更大的范围内推广使用,提高我国生物样本库资源综合利用效率和水平。
(3)科学研究数据化。科学研究数据化是充分利用信息技术、促进科技资源交流、汇集与共享、变革科研组织与活动模式、推动科技转型的一个重要手段,它正在引发21世纪科学与工程的变革。在这个信息爆炸的时代,科研活动之间的交流、科研信息的获取和处理,都在发生着许多新的情况和新的问题。因此,最大限度地满足科研机构和人员之间交流与协作的需要,有效共享浩如烟海的信息,是现代信息通信技术给传统科研带来的巨大变革,而“e-Science”的出现正是这种变革催生的新技术手段。
中国科学院有学者指出,“e-Science”的实质就是“科学研究的信息化”,是信息时代中科学研究环境和科学研究活动的典型体现。它不仅包括采用最新的信息技术,如Grid等,建设起来的新一代的信息基础设施,更有在这种基础设施和相关支撑技术构成的平台上开发的科学研究的应用,以及科学家们在这样一个前所未有的环境中进行的科学研究活动。“e-Science”的实现将为科学家们提供一个信息化的科学研究环境,改变他们从事科学研究活动的方法和手段,甚至直接影响到一些学科的发展。
“e-Science”给科学研究活动所带来的变化是前所未有的、革命性的,其可能产生的深远影响也是绝对不容忽视的。最重要的一点就是“e-Science”使得一种崭新的从事科研活动的方法和模式成为可能,这包括全球性的、跨学科的、大规模科研合作,跨越时间、空间、物理障碍的资源共享与协同工作等等。可以预见,如果e-Science能够实现,那么对于科研信息在整个科学界的充分共享,缩小科学研究领域的数字鸿沟,加速发展中国家的科技进步,以及人类科学研究的更快发展,将具有划时代的意义。因此,也可以预判,e-Science对于研究型医院科学研究的跨越式发展具有特别的意义。
(4)科研管理数据化。科研管理是将基础研究和应用研究的结果与国家和社会需求相结合,并对其进行引导、规划和控制的综合性工作。研究型医院具有开展科学研究、知识创新和科学成果的转化职能。研究型医院的科研管理即针对研究型医院职能对医院内部各种资源进行合理配置,优化组织,协调控制,达到科学成果产出和转化的效率最大化。
科研管理数据化是应用于各个科研院所及高校等研究机构进行科研项目管理、科研成果管理及绩效考核管理等全方位科研管理的一套信息化体系。科研管理数据化带来了管理观念的革新,它突破了条条框框的旧有科研管理模式,而将整个组织的资源、信息有机地组织在一起,不仅可以拓宽管理者的视野以及管理工作的深度和广度,而且也可以增强广大科技工作者之间的交流和理解,从而可以激发他们的创新精神。
(5)临床科研专病数据库。面向临床科研项目管理、数据采集和统计分析的临床专病数据库建设,内容涵盖临床科研中科研项目管理、病例报告表单设计、科研数据采集、查询和导出等主要过程,支持临床科研的回顾性和前瞻性两种类型的研究。实现对整个医疗机构的临床科研的集中统一管理以及单个科研项目的个性化支持;基于规范的临床试验业务流程开发;支持针对不同科研项目的需要,自行定义数据采集,支持多种数据录入方式和数据质量校验;支持从HIS、EMRS、LIS和RIS等第三方系统中自动化采集数据,不需用户重复查找和录入,极大地减轻了科研工作量,更能充分利用医院既有信息系统中的数据,避免产生临床科研的“信息孤岛”。针对不同学科制订不同的CRF,符合目前国内针对重大疾病、慢性病等课题收集样本的习惯,集成流行病调查、检查检验、随访等多种信息,极大地丰富了样本的附加信息,避免了纸质病例报告表错填、漏填、不规范的缺点,方便进行数据的统计、分析。
2013年美国医疗行业八大信息技术预测中,大数据的Ⅰ临床数据分析和“有意义地”使用数据位列其中。通过研究建立临床数据中心重新规划临床研究中数据的获取和应用方式,运用I临床分析,医生能发现最普遍的疾病和状况、不同治疗过程的康复率。就医院而言,也为其提供了运用数据发现罹患慢性病如糖尿病、哮喘和高血压患者的方式。临床分析提供的信息帮助更好地应对疾患,降低昂贵的就诊随访费用,为转化医学研究提供强有力的数据支撑,更有助于提升医疗质量。对医疗机构来说,临床分析都是第一要务,海量数据正开始从研究步入主流。有意义使用数据(meaningful use)是所有医疗机构电子健康档案的应用达到以下要求:改善质量、安全、效率,降低健康差异;将患者和其家庭成员纳入健康管理;改善医疗服务合作;改善人口与公共健康;维护隐私和安全。按要求,医疗机构须实现让患者在36小时内能够浏览住院信息并下载相关数据。只有真正让这些数据得到最有意义的使用,才能将临床问题与实验室研究更好地结合,从而促进科研成果向临床应用的转化。
(五)医疗数据交换平台
1.基于HL7的医疗数据交换平台应用 HL7已经逐渐发展成为了一个医疗数据交换与共享的国际标准,在发达国家得到了广泛推广和应用。如美国的资深医疗信息产品厂商InterSystems公司发布的 Health Share产品,就是基于HL7标准设计的医疗信息交换平台(Health Information Exchange Platform),该平台可以实现全美国范围内的医疗信息的安全交换(secure health information exchange,SHIE),美国罗得岛州、长岛患者信息交换中心(LIPIX)等都采用了Health Share系统为其医疗卫生信息交换提供服务。
在我国,由于历史的原因,国内医疗信息系统在建设初期大都缺乏良好的框架和详细的战略规划,导致目前国内的大多数医院信息系统之间的硬件平台和数据结构都不一样,无法进行跨区域、跨部门、跨行业的医疗数据共享和交换。我国从2000年加入了HL7组织,从那时才逐步开始重视HL7标准的推广和本地化工作,经过了几年的努力,在国内医疗机构、科研单位、高校和相关厂商的大力推动下,HL7才逐步得到人们的认识,取得了一些基于HL7的医疗数据交换的研究成果和少量基于HL7标准的产品。虽然国内对HL7的研究和应用还处在起步阶段,但是随着研究型医院数据化建设的推进,将会有越来越多的医疗信息系统面临医疗数据交换和整合的需求,同时也将会有越来越多的部件和系统采用HL7标准,这将大大推动HL7标准在医疗信息系统数据交换中的发展。
2.数据交换平台的网络结构 数据交换中心负责维护一个基本的医学信息标准库,管理患者索引、电子病历结构树、病历摘要等信息,并为各网关系统提供注册和信息检索等服务,采用ID号作为患者的唯一标识(图13-14)。网关主要负责 HL7标准的消息格式和内部系统之间的转化工作,以及消息的路由工作。为满足各个业务系统对信息标准的需要,网关从数据交换中心读取最新的医学信息标准,数据交换中心负责管理各网关信息和各网关下面所连接的系统信息。网关还要从数据交换中心读取电子病历信息结构库,在聚合电子病历的时候,网关要把各医疗业务系统检测的实时数据变为患者病历摘要信息,保存在交换中心。

图13-14 数据交换平台的网络结构
3.基于HL7协议的数据交换平台架构 基于HL7协议的数据交换平台可采用HL7应用层、Web Services基础架构层和传输层三层架构进行设计,医疗数据以HL7临床文档架构(Clinical Document Architecture,CDA)为载体,封装在HL7消息中进行传输。对于具有特殊的二进制格式大量的医学影像数据和图片,可以通过带附件的SOAP规范来实现传送,从而实现各类医疗数据在数据交换平台上自由交换。
现有医院A电子病历内容转成HL7标准,可以通过调用数据交换平台的WebService方式将HL7消息传输到数据交换平台,格式采用XML,可以保证所有信息交互系统的调用和数据交换。Web Services层通过SOAP协议来实现,以超文本传输协议HTTP作为数据传输的方式,以XML格式描述数据内容。医院B接收HL7标准的信息后,解析XML文档即可获取所需要的信息(图13-15)。

图13-15 数据交换平台架构
免责声明:以上内容源自网络,版权归原作者所有,如有侵犯您的原创版权请告知,我们将尽快删除相关内容。
















