第一节 生物遗传的物质基础
在人们居住的地球上,有大约1000万种生物。生命的奇迹之一是能代代相传。通过对最高等生物——人类的不断探索,终于了解了生命是通过遗传信息的传递而延续的。遗传信息的基本单位即基因,基因存在于细胞染色体的DNA中,一个生物所有遗传信息的总和构成了基因组。在遗传信息的传代中,DNA形成两个拷贝,也就是DNA的复制。
一、基因的结构与功能
从简单的病毒到复杂的高等动、植物细胞,RNA和蛋白质的结构信息都是以基因的形式贮存在DNA(部分病毒是RNA)中的。基因(gene),原称为遗传因子,从分子生物学意义上说,基因是核酸分子中贮存遗传信息的遗传单位,是指贮存有功能的蛋白质多肽链或RNA序列信息及表达这些信息所必需的全部核苷酸序列。基因的基本功能就是贮存、传递和表达遗传信息,并可发生突变,从而决定生物体的性状,甚至导致疾病。因此,基因作为分子医学研究领域的重要内容之一,将分子生物学、遗传学、细胞生物学、医学等多门学科融合到一起,成为人们揭示生命奥妙的重要环节。
1.发展历史 1865年,Mendel在他豌豆杂交试验的基础上提出了生物体内有某种遗传颗粒或基本遗传单位,称为“遗传因子”(genetic factor)。这些遗传因子可以从亲代传递给子代,并决定生物性状。Mendel的遗传规律在35年后得到另外3位植物学家的再次证实,并经Bateson的系统介绍,才被人们所认识。1902~1903年,Sutton和Boveri提出了染色体遗传学(theory of chromosomal inheritance),即认为染色体是“遗传因子”的携带者。1905年,Johannsen首先使用“基因”一词代替“遗传因子”,并提出了基因型(gentoype)和表型(phenotype)的概念。1910年,Morgan首先提出基因定位于染色体上的论点。1944年Avery等人的工作证实了DNA是携带遗传信息和构成染色体的生物大分子。1952年,Hershey和Chase利用噬菌体证实了DNA的遗传性质。
最早阐明基因作用原理的是1945年Beedle和Tatum提出的“一个基因一种酶”的假说。后来,这一假说在许多蛋白质中得到了验证,并被修正为“一个基因一条多肽链”的学说。1953年,Watson和Crick在前人工作的基础上建立DNA双螺旋结构模型,遗传学进入分子水平,标志着现代分子生物学的诞生,基因结构及功能的研究进入了一个新的阶段。1958年Meselon和Stahl提出了DNA半保留复制,1961年Brenner等发现了mRNA,1965年Nirenberg和Khorana破译了密码子,因此20世纪60年代建立了较为完整的分子生物学理论体系——中心法则(central dogma)。20世纪70年代反转录酶的发现进一步完善了中心法则。以后基因工程(Boyer和Cohen,1973)、测序技术(Maxam-Gilbert和Sanger,1977)、PCR(Mullins,1985)等现代分子生物学技术的发展,不仅使染色体的基因定位及物理图谱制作变得轻而易举,而且能详尽地知道定位于染色体DNA上的基因核苷酸顺序。随着2003年人类基因组计划的完成和基因表达调控研究的进展,对基因的了解也不断加深。
20世纪90年代的研究,使Fire和Mello于1998年发现了RNA干扰。近年来,其他非编码RNA(ncRNA)及其功能的不断发现以及表观遗传学的发展,进一步丰富了分子生物学理论,也让人们意识到生命的复杂性远远超出了人们的想象。同时朊病毒和DNA遗传信息的可编排性的发现,出现了DNA遗传、表观遗传和遗传框架之说。对基因的研究也进入了后基因组时代,出现了基因组学、蛋白质组学、系统生物学、生物信息学和整体医学等。这也正应验了那句老话,“科学的发展是永无止境的”。
2.真核生物基因结构 组成一个高等真核生物基因的DNA成分包括:①编码初级转录物的全部顺序;②为正确启动转录及进行转录物加工所必需的最低要求的DNA顺序;③调节转录速率所必需的DNA顺序,如涉及转录的诱导与抑制的顺序以及组织与发育专一性表达的顺序。
现代真核细胞编码蛋白质基因的结构如图2-2所示,包括几个重要元件:转录单位内的外显子和内含子,以及对基因表达和调控具有重要作用的启动子、增强子、终止子等。
(1)转录单位(transcription unit):又称编码区。真核生物的结构基因的编码区DNA序列由编码序列和非编码序列两部分组成,编码序列是不连续的,被非编码序列分割开来,称为断裂基因(split gene)。在结构基因中,编码序列称为外显子(exon),表达多肽链部分。非编码序列称为内含子(intron),又称插入序列。在每个外显子和内含子的接头区都是一段高度保守的共有序列,内含子的5′端是GT,3′端是AG,这种接头方式称为GT-AG法则,普遍存在于真核生物中,是RNA剪接的识别信号。
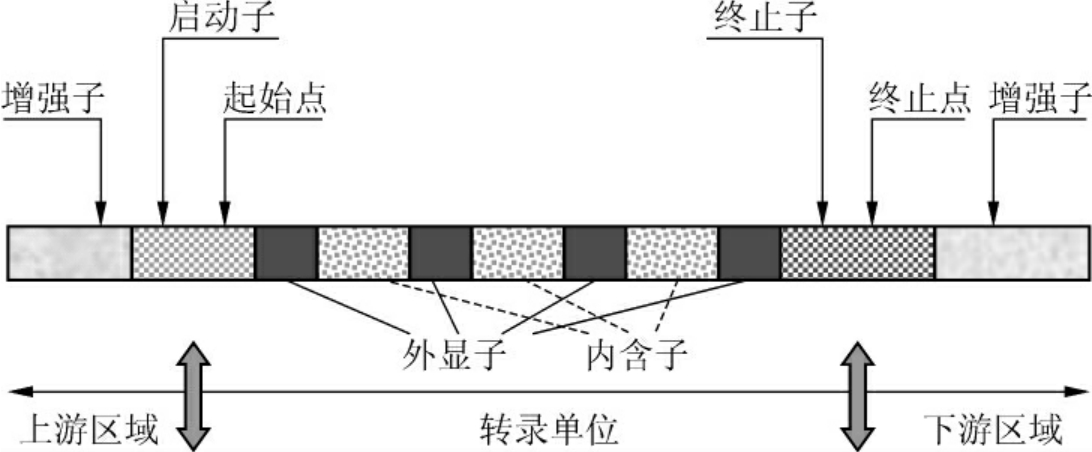
图2-2 现代真核细胞编码蛋白质基因的结构
真核生物内含子和外显子不是完全固定不变的,有时同一DNA链上的某一段DNA序列,当它作为编码某一多肽链的基因时是外显子,而作为编码另一多肽链时,则是内含子。这样,同一基因却可以转录两种或两种以上的mRNA。真核生物某些结构基因没有内含子,如组蛋白基因,干扰素基因等。它们多以基因簇形式存在,大多数的酵母结构基因也没有内含子。
真核基因的外显子与内含子的顺序组成有以下一些特点:①基因组内由内含子分隔的各个外显子的排列顺序与成熟mRNA中对应的外显子顺序保持一致;②断裂基因在个体所有组织细胞中,不论表达与否,其结构不变;③绝大多数内含子都含有3种可能序框的终止密码,当内含子未被切除时,翻译常常在内含子内终止,产生残缺的多肽链;④不同种属的同一基因中,外显子的顺序比较保守,而内含子的顺序变异较大;⑤外显子的长度一般<300bp(碱基对,base pair),内含子的长度较大,可达50~60kb,甚至更长。
(2)启动子(promotor):为RNA聚合酶特异识别和结合的DNA序列,具有方向性,一般位于转录起始点上游的100~200bp范围内,启动子本身不被转录。人类许多基因的转录起始点上游25~30bp的位置常有一段保守序列:TATAAAA或TATATAT,称为TATA盒(TATA box)。典型启动子转录起始点上游-70~-80bp位置常还有一段保守序列:GGC(T)CAATCT,称为CAAT盒,常称为上游启动子元件。有一些基因没有TATA盒与CAAT盒,但在起始点上游-35bp的位置含GGGCGG序列,称为GC盒,发现于一些管家基因中。还有一些基因的启动子既没有TATA盒,也没有GC盒,它们的转录活性很低或没有转录活性,只是在细胞分化、胚胎发育和再生过程中受到调节而活化。
(3)增强子(enhancer):是一段可以位于基因中任何位置的DNA序列,可以与特定转录因子结合而增强基因的转录,它可以位于基因的上游、下游,甚至内含子内。增强子无方向性,在任意位置都有效,但具有组织特异性。后来发现干扰素基因转录时,其增强子序列内含有负调控序列,称为负增强子,又称为沉默子(silencer)。由于负增强子的发现,有人建议用调变子(modulator)取代增强子的概念。
(4)终止子(terminator):基因模板链上的5′端还有终止信号,使RNA聚合酶在模板的特定部位停止作用,终止RNA的合成。无论原核细胞或真核细胞的DNA模板,其终止信号附近都是一个连续的AT碱基对序列区,紧挨着此区的上游则有两段由多个G、C组成的所谓“断裂反向重复区”(interrupted inverted repeat)。通过这两个区域转录出来的RNA链,其碱基能自身配对互补而自动形成发夹式结构,阻止RNA聚合酶的前行。此外,5′端的AT富含区中一连串的A转录出来的RNA链的3′端为一连串的U。凡是具备了这些结构特点的新生RNA即能自动脱离RNA聚合酶和DNA模板,而使RNA的转录终止(图2-3)。
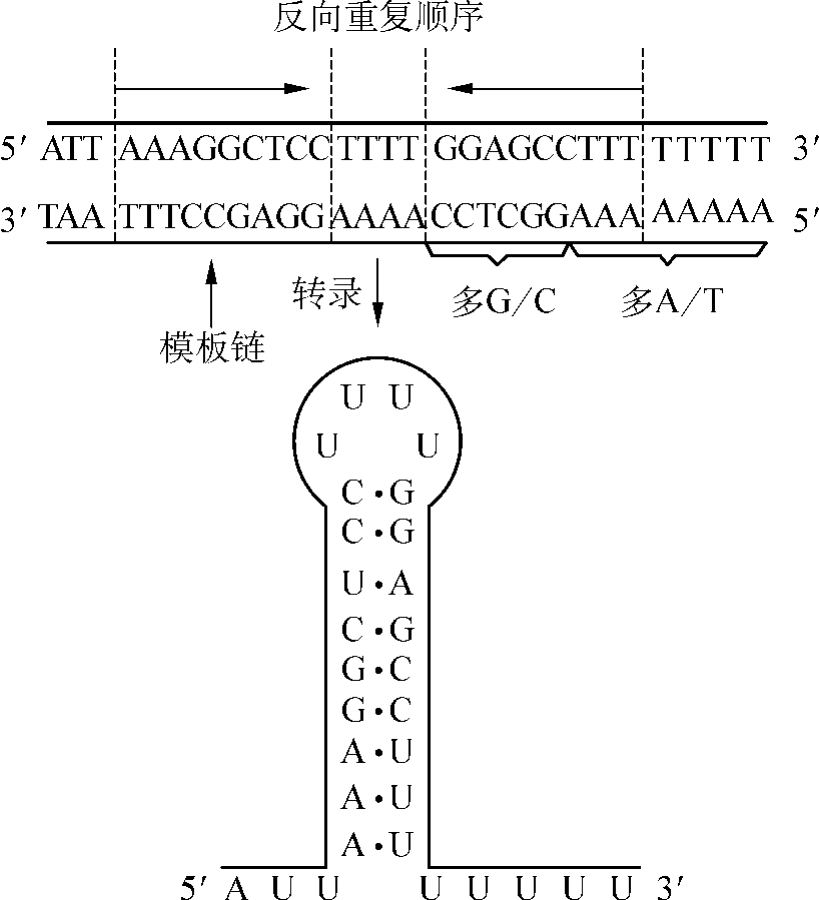
图2-3 终止子结构
二、基因组
基因组(genome),指细胞或生物体中,一套完整单倍体的遗传物质的总和。人类基因组包含核基因组(23条染色体)和线粒体基因组(线粒体上的遗传物质)。真核生物的基因组一般比较庞大,例如人的单倍体基因组由3.2×109bp组成,按1000个碱基编码一种蛋白质计,理论上可有300万个基因。但实际上,人细胞中所含基因总数大概不会超过10万个,根据人类基因组测序结果推测,大约有3.4万个基因,甚至更少。这就说明在人细胞基因组中有许多DNA序列并不转录成mRNA用于指导蛋白质的合成。DNA的复性动力学研究发现这些非编码区往往都是一些大量的重复序列,这些重复序列或集中成簇,或分散在基因之间。在基因内部也有许多能转录但不翻译的间隔序列(内含子)。因此,在人细胞的整个基因组当中只有很少一部分(约占2%)的DNA序列用以编码蛋白质(图2-4)。
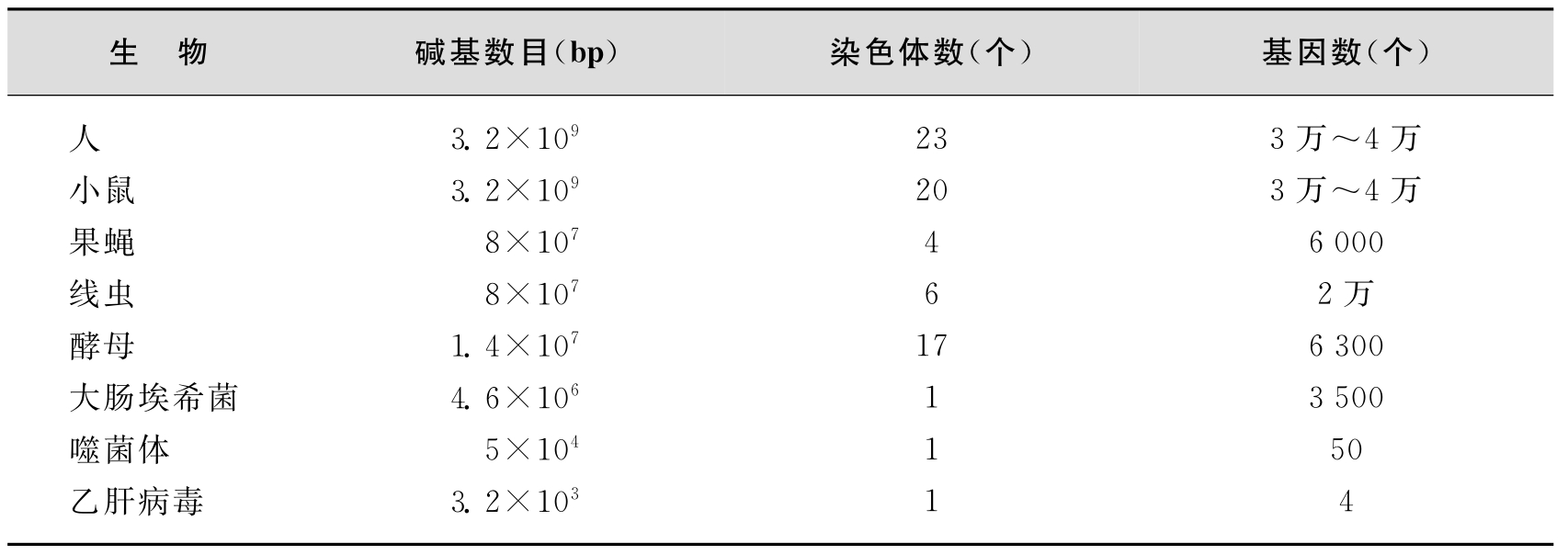
简而言之,真核生物基因组有以下特点:①真核生物基因组DNA与蛋白质结合形成染色体,储存于细胞核内,除配子细胞外,体细胞内的基因组是双份的(即双倍体,diploid),即有两份同源的基因组;②真核细胞基因转录产物多为单顺反子,一个结构基因经过转录和翻译生成一个mRNA分子和一条多肽链;③存在大量重复序列,且大部分为非编码序列;④基因组中不编码的区域多于编码区域;⑤大部分基因含有内含子,因此,基因是不连续的;⑥基因组远远大于原核生物的基因组,具有许多复制起点,而每个复制子的长度较小。为了便于比较,表2-1列出了部分生物的染色体数和基因数目。

图2-4 人类基因组组成
表2-1 部分生物基因组中正常染色体数及基因数
1.重复序列 真核基因组的一大特点就是存在大量的重复序列,除了编码rRNA、tRNA、组蛋白和免疫球蛋白的结构基因外,大部分的重复序列为非编码序列。一般根据重复基本单位的重复次数的多少,可将重复序列分成三类,即高度重复序列、中度重复序列和单拷贝序列;根据重复序列的结构特点又可分为反向重复序列、串联重复序列和散在重复序列。
(1)反向重复序列(inverted repetitive sequences):由两个相同顺序的互补拷贝在同一DNA链上反向排列而成。变性后再复性时,同一条链内的互补的拷贝可以形成链内碱基配对,形成发夹式或“+”字形结构。反向重复可有一到几个核苷酸的间隔,也可以没有间隔。没有间隔的又称为回文结构(palimdrome)。
(2)串联重复序列(tanden repeats):是以一个重复单位头尾相连形成的,人类基因组中的串联重复序列有大卫星DNA(macrosatellite DNA)、小卫星DNA(minisatellite DNA)和微卫星DNA(microsatellite DNA)。大卫星DNA属于高度重复序列的串联重复序列,也称为经典卫星DNA,是由2~10bp组成,成串排列,在基因组中重复频率高,可达百万(106)以上。由于这类序列的碱基组成不同于其他部分,可用等密度梯度离心法将其与主体DNA分开,因而称为卫星DNA或随体DNA。在人基因组中卫星DNA占5%~6%。按照它们的浮力密度不同,人的卫星DNA可分为Ⅰ、Ⅱ、Ⅲ、Ⅳ4种。卫星DNA还有另一些分类的名称,如α卫星DNA是灵长类特有的单元为171bp的高度重复序列,最初是在非洲青猴基因组中发现,现在已确定分布于人染色体的着丝粒区。β卫星DNA家族是单元为68bp的串联重复序列,富含GC。γ卫星DNA是220bp的串联重复。
小卫星DNA是由中等大小的串联重复序列构成,一般为0.1~20kb长,每个重复单位在15~70bp。与卫星DNA不同,小卫星DNA在染色体上分布于常染色质区和端粒处,可分为具有GGGCGGAXG核心序列的高度可变的小卫星DNA和具有TTAGGG重复序列的端粒DNA。小卫星DNA重复单位的拷贝数在群体内存在非常大的变异,有着高度的多态性。DNA指纹就是利用这一特性来区别同一种属中不同的个体。这项技术发展很快,目前在法医学、分类学、生态学及动、植物遗传育种等领域有广泛的应用。
进入20世纪90年代以来,人们发现有些串状重复序列的重复单位仅仅有2~5bp,如(CA)n、(GT)n、(GAA)n等。由于重复单位的长度比小卫星更短,故称为微卫星DNA,又称为短小串联重复序列(short tandem repeats,STRs)或简单重复序列(simple sequence repeats,SSRs)。它们分布于染色体的常染色质区,多位于编码区附近,也可位于内含子、启动子、Alu序列中。微卫星DNA的生物学功能尚不清楚,可能对基因的调节起作用,也可能是重组的热点所在。微卫星DNA约占真核基因组的5%,在人类基因组中有(5×104)~(1×105)个(CA)n重复序列。每个微卫星DNA序列的基本单元重复次数在不同基因型间差异很大,从而形成其座位的多态性,并按孟德尔共显性方式在人群中世代相传。因此,这种多态性标志已广泛用于构建人类遗传连锁图谱、基因定位、遗传病诊断、肿瘤细胞染色体分离与重组以及亲子鉴定等法医学检查。
(3)散在重复序列:是指重复单位并不相连,而是散布在整个基因组中的序列。散在重复序列大致分为4种类型:LTR元件、长散在核元件、短散在核元件和DNA转座子,但主要以长散在核元件、短散在核元件形式出现。
1)短散在核元件(short interspersed nuclear elements,SINES):这类重复单位的平均长度约为300bp,一般<500bp,它们与平均长度约为1000bp的单拷贝顺序间隔排列。重复次数可达105左右。如Alu家族,Hinf家族等属于这种类型的中度重复序列。
2)长散在核元件(long interspersed nuclear elements,LINES):这类重复顺序的长度>1000bp,平均长度为3500~5000bp,它们与平均长度为13000bp(个别长几万bp)的单拷贝顺序间隔排列。也有的实验显示人基因组中所有LINES之间的平均距离为2.2kb,重复次数一般在1×14左右,如KpnⅠ家族等。
(4)单拷贝序列(single copy sequences)或低度重复顺序:在单倍体基因组中只出现一次或数次,因而复性速度很慢。单拷贝顺序在基因组中占50%~80%,如人基因组中有60%~65%的顺序属于这一类。在绝大多数真核生物的单倍体基因组中,编码蛋白质的结构基因都不重复,是单拷贝的基因。单拷贝顺序中储存了巨大的遗传信息,编码各种不同功能的蛋白质。目前尚不清楚单拷贝基因的确切数量,但已经清楚的是在单拷贝顺序中只有一小部分用来编码各种蛋白质,其余部分的功能尚不清楚。
2.基因家族与假基因 基因家族(gene family)是指核苷酸序列和编码产物的结构具有一定程度同源性的一组基因。这些由某一祖先基因经过重复和变异所产生的一组基因,常又称为多基因家族(multi-gene family)。根据基因家族内各成员同源性的程度不同可分为以下几种:①如rRNA、tRNA和组蛋白等序列相同的基因家族;②如人生长激素(hGH)、胚胎促乳素(HCS)和催乳素基因核酸序列高度同源的家族;③编码产物具有同源功能区的src家族;④如具有天谷丙天(DEAD)盒基因家族,编码产物具有8个氨基酸保守基序。由多基因家族及单基因组成的更大的基因家族,称为基因超家族(gene superfamily),它们在结构上有一定程度的同源性,但功能并不一定相同。在多基因家族中,某些成员并不产生有功能的基因产物,这些基因称为假基因(pseudo-gene)。假基因与有功能的基因同源,原来可能也是有功能的基因,但由于缺失、倒位或点突变等,使这一基因失去活性,成为无功能基因。
3.端粒 线性真核基因组DNA的末端有一种特殊的短而简单的重复序列,称为端粒(telomere)。端粒的DNA序列相当保守,由5~8bp的重复单位串联而成,重复次数在不同的生物中变化较大,如小鼠的端粒DNA序列长150kb,而人端粒DNA序列长4~15kb,由5′-TTAGGG-3′的重复序列构成,属于小卫星DNA中的一种。端粒在DNA复制时,由端粒酶(telomerase)来完成复制,以此来保证DNA的长度不会因复制而变短,保护了DNA链的完整复制。人类的端粒酶是由端粒酶RNA(TR)、端粒酶相关蛋白和端粒酶催化亚基(TERT)3个部分组成。端粒酶以RNA为模板,在TERT的作用下催化合成端粒DNA。其结构和催化机制都与反转录酶相似,人们因此也把它归属于反转录酶家族。最近研究表明,端粒由DNA和蛋白质复合物组成,其作用主要体现在以下4个方面:①可以使细胞的DNA损伤修复机制正确区分正常的染色体末端和损伤DNA双链的末端;②保证了染色体的完整性;③在基因组的功能方面起调控作用;④参与染色体在细胞核内的定位与组织。端粒末端通常形成高度精密的复合体,防止被核酸酶等物质降解,当端粒末端的高级结构被破坏后,染色体便发生融合,这样在细胞进行有丝分裂时染色体就会断裂,造成基因组的不稳定性。因此,端粒与细胞癌变、细胞的衰老和寿命的长短密切相关。
4.转座子 在真核生物基因组中,编码序列在染色体中的位置相对比较稳定,但也有一些中度重复序列往往可在染色体间或基因间移动。这些存在于DNA上可自主复制和移位的基本单位称为转座子(transposon)。目前认为,多数生物体有自发突变且有重要表型效应出现的原因,可源于转座子的可动性,并且可以导致宿主基因组发生从点突变到染色体重排的一系列变化。在果蝇中已发现30多种可移动成分。在人类中多存在反转录转座子(retrotransposon),反转录转座子不同于转座子,是以DNA→RNA→DNA的途径来实现转座的,在整合酶的作用下由RNA反转录生成的、以DNA状态存在的反转录转座子整合到宿主基因组中。这样,反转录转座子在宿主基因组中的拷贝数得到不断积累,从而使基因组增大。由于反转录转座子带有增强子、启动子等调控元件,所以会影响宿主基因的表达,在生物进化过程中反转录转座子起着不可忽视的作用。
5.线粒体基因组 线粒体是生物氧化的场所,呼吸链中的某些蛋白质或酶的编码基因就在mtDNA上。线粒体还能独立合成一些蛋白质,因为线粒体有自己的rRNA、tRNA和核糖体等可以用来表达自己的基因。
动物的线粒体基因组是共价闭合的双链DNA(mtDNA),一般长度为15.7~19.5kb核酸序列和组成比较保守。根据碱性氯化铯密度梯度离心中双链密度不同分为重链(H链)和轻链(L链)。哺乳动物mtDNA中除一个蛋白质基因(ND6)和8个tRNA基因由L链编码外,其余的大部分基因都由H链编码。人的mtDNA由16569个碱基组成,果蝇由16019个碱基组成。现已知线粒体的基因组至少含有13个蛋白质基因、22个tRNA基因和2个rRNA基因。蛋白基因包括细胞色素b基因(Cytb)、细胞色素氧化酶3个亚基基因(COXⅠ、COXⅡ、COXⅢ)、NADH氧化还原酶7个亚基基因(ND1、ND2、ND3、ND4、ND4L、ND5、ND6)和ATP酶2个亚基基因(ATPase6、ATPase8),这13个蛋白或亚基都是线粒体内膜呼吸链的组分。另外,还有一些抗药性基因也在mtDNA上。哺乳动物mtDNA基因组上基因之间无间隔区(spacer),基因中亦无内含子,甚至有基因重叠现象。
三、染色体
人单倍体细胞基因组含有3.2×109bp,若将所有染色体相连并充分伸展的话,其长度可达2m左右。如此巨大的DNA链要全部贮存在小小的细胞核染色体中,必然存在着适宜的包装形式。此外,染色体中还含有大量的蛋白质和有限的RNA,它们与DNA共同构成十分紧密的复杂结构。这种结构不仅能满足贮存DNA的数量要求,而且还应该有利于其功能的实现。
1.染色体的化学组成 DNA是染色体的主要化学成分,也是遗传信息的载体。此外,在认识DNA重要性之前,已经知道染色体还含有大量的蛋白质,后来又发现了RNA。生化研究表明:上述三类组成染色体的化学成分中,蛋白质含量约为DNA的2倍,根据组成蛋白质的氨基酸特点分为组蛋白和非组蛋白两类。RNA含量很少,还不到DNA量的10%。
(1)组蛋白(histones):是指染色体中的碱性蛋白质,其特点是富含两种碱性氨基酸(赖氨酸和精氨酸),碱性和酸性氨基酸之比为1.4~2.5,等电点(pI)为7.5~10.5。根据赖氨酸和精氨酸在蛋白质分子中的相对比例,又将组蛋白分为5种小类型,即组蛋白H1、H2、H3、H4和H5。H1极富赖氨酸,H2A和H2B稍富赖氨酸,H3和H4富含精氨酸。这5种组蛋白的氨基酸全顺序均已确定。在各种物种中,H3和H4的序列极少有差异,这种生物进化上的高度保守性预示着其功能的重要性。其他3种组蛋白在不同种属之间存在着较大的差异。所有真核生物染色体中均含有这五种组蛋白。组蛋白的含量与DNA含量之比约为1∶1。组蛋白中这些强极性氨基酸使蛋白质带上大量电荷,成为组蛋白与DNA结合及蛋白质之间相互作用的主要化学力之一。
(2)非组蛋白(non-histone protein,NHP):是指染色体中组蛋白以外的其他蛋白质,它是种类繁杂的各种蛋白质的总称。估计总数为300~600,分子量为7000~80000,等电点为3.9~9.2。对于其中许多单个成分的结构和功能,目前还了解甚少。已经知道,一些非组蛋白与基因表达及染色体高级结构的维持有关。参与基因复制、转录及核酸修饰的酶类(如各种DNA和RNA聚合酶等)就是一类重要的非组蛋白。另外,非组蛋白中还包括一类高迁移率组(high mobility group,HMG)蛋白质,此类蛋白质因在凝胶电泳上泳动速度快而得名。已经明确其中的一些蛋白质(如HMG14和HMG17)在转录活性区含量丰富,可以认为是参与转录调控的蛋白质。
2.核小体的结构 通过电子显微镜观察破裂的间期细胞流出的染色质,可以看到染色质纤维呈非连续性颗粒状,就像一条细线上串联着许多有一定间隔的小珠,这些小珠状颗粒被称为核小体(nucleosomes),由Kornberg于1974年发现。核小体由核心颗粒(core particle)和连接区DNA(linker DNA)两部分组成,146bp的DNA片段和H2A、H2B、H3和H4各两分子组成核心颗粒(图2-5),而60bp左右连接区DNA则与H1结合。DNA与组蛋白八聚体的这种相互作用,对保护DNA链免受细胞核中核酸酶的消化十分重要。
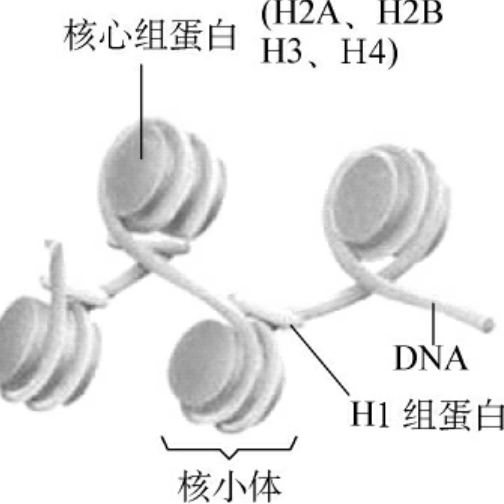
图2-5 核小体结构
3.染色体的包装 实际上是指细胞核DNA在双螺旋基础上的进一步结构变化,这些结构变化总的看是更高层次的超螺旋形成。上面讨论的核小体,可视为染色体DNA的一级包装,即由直径2nm的DNA双螺旋链绕组蛋白形成直径11nm的核小体“串珠”结构。若增大离子强度,并保留H1,通过电镜可观察到10nm纤维会折转成较粗的30nm纤维,这种纤维即染色体DNA的二级包装,目前较公认的二级包装结构模型是螺线管纤维(solenoid fiber)。它是由核小体纤维盘绕形成的一种中空螺线管,其外径为30nm,每圈含6个核小体。螺线管纤维基础上的更高一级包装是形成环状螺线管,这种结构是30nm纤维缠绕在一个由某些非组蛋白构成的中心轴(central axis)骨架上形成的。即螺线管纤维相隔一定间距的某些区段被“拉拢”固定在蛋白轴上,从而产生了许多从骨架上伸出的纤维环(loops)。螺线管纤维需进一步以某种方式盘绕、折叠,最终完成细胞生长和繁殖的不同时期的染色体包装。这种在更高层次上的复杂的包装是以何种方式进行的,目前尚无明确定论。从螺线管纤维环到包装形成染色体,应该是DNA压缩程度最高的阶段,估计在200~240倍。经各级包装后染色体DNA总共被压缩了数千倍,这样,才能使每个染色体中几厘米长的DNA分子容纳在直径数微米(如人细胞核的直径为6~7μm)的细胞核中。
四、DNA复制及损伤修复
(一)DNA复制
体细胞有丝分裂的间期,在DNA聚合酶的作用下,以一个亲代DNA分子的两条链为模板,合成两个结构上完全相同的子代DNA分子的过程称为DNA复制。1953年,Watson和Crick在提出DNA双螺旋模型时就指出,由于互补碱基的配对原则,在DNA复制时,只要双链DNA解开,以每一条链为模板以合成其互补链,即可形成两个与亲代完全一样的DNA分子。在此过程中,每个子代DNA的一条链来自亲代DNA,另一条链则是新合成的。这种复制方式称为半保留复制(semi conservation replication)。1958年,Meselson和Stanl采用含15N的NH4Cl培养大肠埃希菌,再放入于以14N为氮源的普通培养液中繁殖,然后用CsCl密度离心方法测定分裂时DNA复制后的密度变化,证实了DNA的半保留复制机制。通过DNA分子的复制,把亲代的遗传信息传给子代,从而使得前后代保持了一定的连续性。
DNA复制的基本过程正如Watson和Crick所预测的那样,但是在DNA复制中涉及了许多细胞成分。DNA的复制过程大体上可以分为复制的启动、复制的延伸和复制的终止3个阶段。DNA复制过程极其复杂,至今对其详细过程的了解还很不全面,多数资料来源于原核生物。近几年,对于真核生物的DNA复制也有所认识。真核生物的DNA复制基本上与原核生物相似,但更为复杂。
1.起始点 DNA复制在生物细胞中要从DNA分子上特定位置开始。这个特定的位置就称为复制起点(origin of replication),用ori表示。DNA复制从起点开始双向进行直到终点为止,每一个这样的DNA单位称为复制子或复制单元。在原核生物中每个DNA分子上就有一个复制子;而在真核生物中每个DNA分子有许多个复制子,每个复制子长50~200kb。因此,真核细胞DNA的复制是由许多个复制子共同完成的,而且形成两个复制叉进行双向复制。DNA复制不是随机的从DNA分子上的任何一点起始,而是从特定的区域开始,这说明复制起点有其结构上的特殊性。分子遗传学者们利用分子克隆技术,分离出大肠埃希菌染色体DNA复制起点oriC。它由422bp的DNA片段组成,其核苷酸序列已经搞清。其结构特点:①在oriC区域内有一系列对称排列的反向重复序列,即回文结构(palindrome),与复制酶系统的识别有关;②在oriC区中还有两个转录启动区(启动子)的核苷酸序列,这暗示了转录可能在大肠埃希菌染色体DNA复制起始中有着重要的作用。目前已有许多实验表明转录确实是复制的起始所必需的,但具体的作用机制尚不清楚。
对人及高等哺乳动物DNA复制起始位点的了解远不如对原核生物及酵母的复制起始位点的了解。1998年,Aladjem等发现在人珠蛋白基因的一个片段中的几个基本位点具有较高的复制起始功能,可能是DNA复制起始位点。
2.复制过程 分为4个阶段。
第一阶段:亲代DNA分子超螺旋的构象变化及双螺旋的解链,将复制的模板展现出来。参与的酶包括拓扑异构酶(DNA topisomerase)、螺旋酶(helicase)、解链酶及单链结合蛋白(single strand DNA binding protein,SSBP)等。
第二阶段:为复制的引发阶段,由复制因子X(n蛋白),复制因子Y(n′蛋白),n″蛋白,i蛋白,DnaB蛋白和DnaC蛋白等6种蛋白质组成的引发前体(preprimosome),在单链DNA结合蛋白的作用下与单链DNA结合生成中间物,这是一种前引发过程。引发前体进一步与引物酶(primase)组装成引发体(primosome)。引发体可以在单链DNA上移动,在DnaB亚基的作用下识别DNA复制起点位置。首先在前导链上由引物酶催化合成一段RNA引物,然后,引发体在滞后链上沿5′→3′方向不停地移动,合成RNA引物。
第三阶段:为DNA链的延长(图2-6),在引物RNA合成基础上,以每条链为模板,按碱基互补配对原则(A∶T,G∶C),由DNA聚合酶催化进行DNA链的5′→3′方向合成,前导链连续地合成出一条长链,随从链合成出冈崎片段。真核生物DNA聚合酶有α、β、γ、δ及ε5种,DNA复制是在DNA聚合酶α与DNA聚合酶δ相互配合下催化进行的,还有一些酶及蛋白质因子参与反应。DNA Polα与引发酶共同起引发作用,然后由DNA Polδ催化前导链及随从链的合成。DNA Polγ是线粒体中DNA复制酶。DNA Polδ及ε均有外切酶活性,校正复制中的错误。它们的5′→3′外切酶活性可能在切除引物RNA中也有作用。去除RNA引物后,片段间形成了空隙,由DNA聚合酶补上。在连接酶作用下,各片段连接成为一条长链。
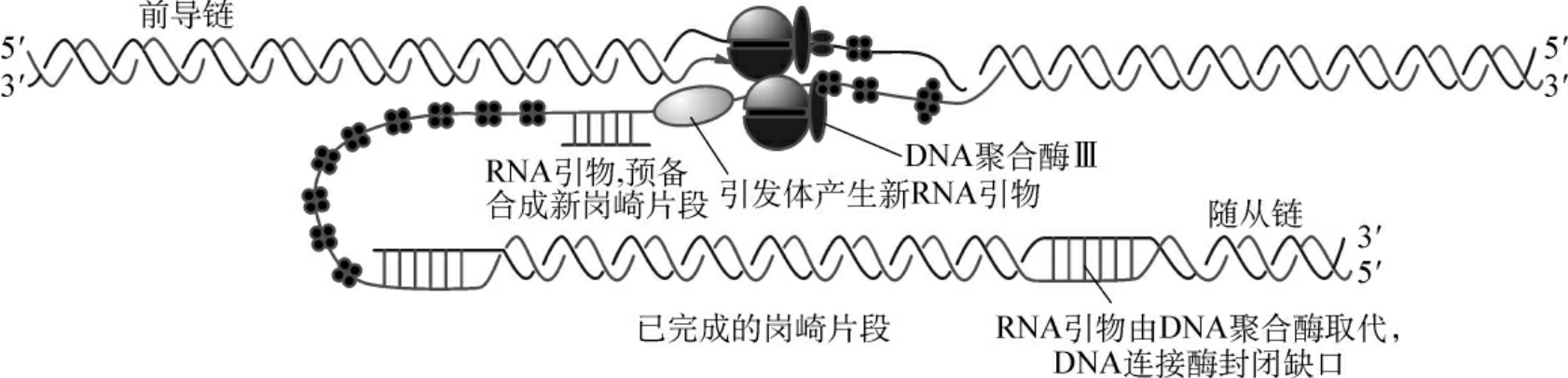
图2-6 DNA链的延伸过程
第四阶段:为终止阶段,复制叉行进到一定部位就停止前进,最后前导链与随从链分别与各自的模板形成两个子代DNA分子,到此复制过程就完成了。过去认为,DNA一旦复制开始,就会将该DNA分子全部复制完毕,才终止其DNA复制。但最近的实验表明,在DNA上也存在着复制终止位点,DNA复制将在复制终止位点处终止,并不一定等全部DNA合成完毕。但目前对复制终止位点的结构和功能了解甚少。
在DNA复制终止阶段令人困惑的一个问题是,线性DNA分子两端是如何完成其复制的?已知DNA复制都要有RNA引物参与。当RNA引物被切除后,中间所遗留的间隙由DNA聚合酶Ⅰ所填充。但是,在线性分子的两端以5′→3′为模板的合成,其末端的RNA引物被切除后是无法被DNA聚合酶所填充的。因为DNA聚合酶不具备从头合成的能力和3′→5′方向的合成能力,这就存在“末端复制问题”。这个问题的解决有赖于端粒和端粒酶的发现,原来DNA末端存在一个重复序列的结构,并通过端粒酶依赖机制和端粒替代延长机制来保证DNA链的完整复制。
(二)DNA损伤及其修复
DNA分子的完整性和稳定性是复制的高度真实性的基础,但生物体内外环境都存在着使DNA分子损伤的因素,引起DNA突变(mutation)。如若不加以纠正,则细胞就会产生错误的表达信息或表达变异的产物,这往往是疾病和衰老发生的原因。但生物体内也有一套有效的纠正机制,以恢复正常DNA结构和功能,称为DNA修复(DNA repair)。
1.DNA损伤和突变的引发因素
(1)DNA分子的自发性损伤:以DNA为模板按碱基配对进行DNA复制是一个严格而精确的事件,但也不是完全不发生错误的。碱基配对的错误频率约为10-1~10-2,经DNA聚合酶校正后的错配率仍约在10-10左右,即每复制1010个核苷酸大概会有一个碱基的错误。此外,体内DNA修复系统在对DNA损伤进行修复时的DNA合成同样也会发生碱基配对错误,造成DNA损伤。
生物体内DNA分子可以由于各种原因发生变化,至少有以下类型:①碱基的异构互变:DNA中的4种碱基各自的异构体间都可以自发地相互变化(如烯醇式与酮式碱基间的互变),这种变化就会使碱基配对间的氢键改变,可使腺嘌呤能配上胞嘧啶、胸腺嘧啶能配上鸟嘌呤等,如果这些配对发生在DNA复制时,就会造成子代DNA序列与亲代DNA不同的错误性损伤;②碱基的脱氨基作用:碱基的环外氨基有时会自发脱落,从而胞嘧啶会变成尿嘧啶、腺嘌呤会变成次黄嘌呤(H)、鸟嘌呤会变成黄嘌呤(X)等,遇到复制时,U与A配对、H和X都与C配对就会导致子代DNA序列的错误变化;③脱嘌呤与脱嘧啶:自发的水解可使嘌呤和嘧啶从DNA链的核糖磷酸骨架上脱落下来;④碱基修饰与链断裂:细胞呼吸的副产物O2-、H2O2等会造成DNA损伤,能产生胸腺嘧啶乙二醇、羟甲基尿嘧啶等碱基修饰物,还可能引起DNA单链断裂等损伤。
(2)物理因素引起的损伤:DNA分子损伤最早就是从研究紫外线的效应开始的。当DNA受到最易被其吸收波长(260nm左右)的紫外线照射时,主要是使同一条DNA链上相邻的嘧啶以共价键连成二聚体,相邻的两个T或两个C,或C与T间都可以连成二聚体,其中最容易形成的是TT二聚体,从而影响DNA的复制。254nm紫外线还可引起DNA链的断裂。
电离辐射损伤DNA有直接和间接的效应,直接效应是DNA直接吸收射线能量而遭损伤,间接效应是指DNA周围其他分子(主要是水分子)吸收射线能量产生具有很高反应活性的自由基进而损伤DNA。电离辐射可导致DNA分子的多种变化,包括DNA链上的碱基氧化修饰、过氧化物的形成、碱基环和脱氧核糖的破坏、DNA链断裂、DNA链交联和DNA-蛋白质交联等。
(3)化学因素引起的损伤:许多化学因素,包括烷化剂、碱基类似物、修饰剂和一些人工合成或环境中存在的化学物质(例如亚硝酸盐、羟胺、黄曲霉素等),这些都是诱发突变的化学物质或致癌剂,使DNA碱基烷基化后错配或脱落断裂。其中双功能烷化剂如氮芥、硫芥等、一些抗癌药物如环磷酰胺、苯丁酸氮芥、丝裂霉素等、某些致癌物如二乙基亚硝胺等,其两个功能基可同时使两处烷基化,结果就能造成DNA链内、DNA链间以及DNA与蛋白质间的交联。而碱基类似物如溴尿嘧啶(5-BU)、氟尿嘧啶(5-FU)、2-氨基腺嘌呤(2-AP)等,由于其结构与正常的碱基相似,进入细胞能替代正常的碱基掺入到DNA链中而干扰DNA复制合成,例如5-BU结构与胸腺嘧啶十分相近,在酮式结构时与A配对,却又更容易成为烯醇式结构与G配对,在DNA复制时导致A-T转换为G-C。
2.DNA损伤、突变的类型及其意义 上述各种因素造成DNA损伤(DNA damage),引起基因突变(gene mutation)。归纳DNA损伤后分子最终的改变,有以下几种类型:①点突变:指DNA上单一碱基的变异。嘌呤替代嘌呤(A与G之间的相互替代)、嘧啶替代嘧啶(C与T之间的替代)称为转换(transition);嘌呤变嘧啶或嘧啶变嘌呤则称为颠换(transvertion)。②缺失:指DNA链上一个或一段核苷酸的消失。③插入:指一个或一段核苷酸插入到DNA链中。在为蛋白质编码的序列中如缺失及插入的核苷酸数不是3的整倍数,则发生读框移动(reading frame shift),使其后所译读的氨基酸序列全部混乱,称为移码突变(frame-shift mutation)。④倒位或转位:指DNA链重组使其中一段核苷酸链方向倒置,或从一处迁移到另一处。⑤双链断裂。
突变或诱变对生物可能产生3种后果:①丧失某些功能甚至致死性,是某些疾病的发病基础;②改变基因型而不改变表现型,形成了个体之间的多态性;③发生了有利于物种生存的结果,使生物进化。
3.DNA损伤的修复 DNA修复(DNA repairing)是细胞对DNA受损伤后的一种反应,这种反应可能使DNA结构恢复原样,重新能执行它原来的功能。但有时并非能完全消除DNA的损伤,只是使细胞能够耐受这DNA的损伤而能继续生存。对不同的DNA损伤,细胞可以有不同的修复反应。
(1)光修复(light repair):是最早发现的DNA修复方式。修复是由细菌中的DNA光复活酶(photolyase)完成,此酶能特异性识别紫外线造成的核酸链上相邻嘧啶二聚体,并与其结合,在光激活下将二聚体分解为两个正常的嘧啶单体,然后酶从DNA链上释放,DNA恢复正常结构。已发现多种生物含有光复活酶,但未发现人类有此类酶。
(2)切除修复(excision repair):是修复DNA损伤最为普遍的方式,对多种DNA损伤包括碱基脱落形成的无碱基位点、嘧啶二聚体、碱基烷基化、单链断裂等都能起修复作用。这种修复方式普遍存在于各种生物细胞中,也是人体细胞主要的DNA修复机制。修复过程需要多种酶的一系列作用(如人体的切除修复需30多种因子的参与),首先由核酸酶识别DNA的损伤位点,在损伤部位的5′侧切开磷酸二酯键,由3′→5′核酸外切酶将有损伤的DNA片段切除。然后在DNA聚合酶的催化下,以完整的互补链为模板,按3′→5′方向合成DNA链,填补已切除的空隙。最后,由DNA连接酶将新合成的DNA片段与原来的DNA断链连接起来。这样完成的修复能使DNA恢复原来的结构。切除修复包括碱基切除修复(base excision repair,BER)和核苷切除修复(nucleotide excision repair,NER)两种形式。
(3)错配修复(mismacth repair,MMR):是一种特殊的切除修复形式。DNA复制时出现的碱基错配可有DNA聚合酶进行纠正,该纠正机制被称为首次纠正机制。但首次纠正机制不能保证修复所有的错配,部分错误碱基可逃脱该纠正机制,遗留在DNA链上。遗留错误碱基的纠正由错配修复系统来完成。DNA错配修复基因首先在细菌和酵母中发现,随后在用连锁分析方法研究HNPCC家系易感基因的过程中,发现了人类的类似基因。涉及人类DNA错配修复的主要有6个蛋白质,即hMSH2、hMSH3、hMSH6、hMLH1、hMLH3、hPMS1。碱基错配后,首先被两种MutS同源蛋白异二聚体复合物MSH2/hMSH6(MutSα)和MSH2/MSH3(MutSβ)识别。其中MutSα识别并结合于碱基-碱基错配的位置,MutSβ识别并结合于较大片段插入/缺失错配的位置。错配碱基被识别后,MMR系统中另外的一种MutL同源蛋白异二聚体MLH1/PMS1就与已结合到错配碱基位置的MutSα或MutSβ相互作用形成一种暂时性的复合物,从而启动错配修复,并在系统其他协同蛋白,如hEXO1等的相互配合下切除含有错配碱基的一段DNA链,合成与母链完全匹配的新链,以代替被切除的DNA链片段,从而完成对含有错配碱基的DNA链的修复。
(4)重组修复(recombinational repair):上述的切除修复在切除损伤段落后是以原来正确的互补链为模板来合成新的段落而做到修复的。但在某些情况下没有互补链可以直接利用,例如在DNA复制进行时发生DNA损伤,此时DNA两条链已经分开,其修复可用以下的DNA重组方式:①受损伤的DNA链复制时,产生的子代DNA在损伤的对应部位出现缺口;②完整的另一条母链DNA与有缺口的子链DNA进行重组交换,将母链DNA上相应的片段填补子链缺口处,而母链DNA出现缺口;③以另一条子链DNA为模板,经DNA聚合酶催化合成一新DNA片段填补母链DNA的缺口,最后由DNA连接酶连接,完成修补(图2-7)。
重组修复不能完全去除损伤,损伤的DNA段落仍然保留在亲代DNA链上,只是重组修复以后新合成的DNA分子是不带有损伤的,但经多次复制后,损伤就被“冲淡”了,在子代细胞中只有一个细胞是带有损伤DNA的。
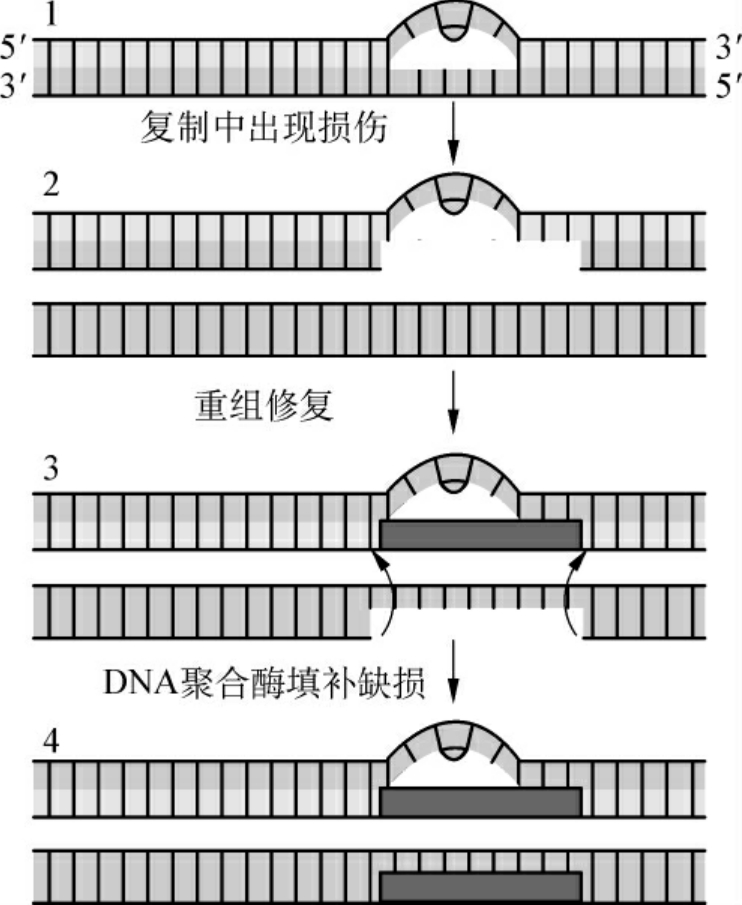
图2-7 重组修复示意图
(5)SOS修复:是指DNA受到严重损伤、细胞处于危急状态时所诱导的一种DNA修复方式。此时,细胞的正常修复机制被抑制,损伤处的DNA链空缺,再由损伤诱导产生的一整套的特殊DNA聚合酶——SOS修复酶类,催化空缺部位DNA的合成。这时补上去的核苷酸几乎是随机的,不能保证序列的正确性,然而还是保持了DNA双链的完整性,使细胞得以生存。但留下的错误较多,故又称为错误倾向修复(error-prone repair),使细胞有较高的突变率。
应该说目前对真核细胞DNA修复的反应类型、参与修复的酶类和修复机制了解还不多,但DNA损伤修复与细胞突变、寿命、衰老、肿瘤发生、辐射效应、某些毒物的作用都有密切的关系。人类遗传性疾病已发现4000多种,其中不少与DNA修复缺陷有关,这些DNA修复缺陷的细胞表现出对辐射和致癌剂的敏感性增加。例如着色性干皮病(xerodermappigmentosum,XP)、运动失调毛细血管扩张症(ataxiatelangiectasia,AT)、遗传性非息肉结肠癌(hereditarynon-polyposiscoloncancer,HN-PCC)和乳腺癌等均与DNA修复缺陷相关。
免责声明:以上内容源自网络,版权归原作者所有,如有侵犯您的原创版权请告知,我们将尽快删除相关内容。

















