第十章 结核分枝杆菌的分子生物学
分枝杆菌菌属是一类最为古老的,具有各式各样独特特性的细菌。它们既可是土壤和水中的自由生长的共栖菌,也可对人类和动物致病。至少有25种分枝杆菌与人类中的疾病有关,其中结核分枝杆菌是人类结核病的病原菌。据估计,1990~1999年全世界约有9 000万新的结核病病例。结核病是成人中由于单一感染因子所致死的首要原因。如果全球结核病的控制仍停留在1990年的水平,则至2000年将有3 000万人死于这一疾病。此外结核病尚可与人类免疫缺陷病毒(HIV)发生共同感染,而且对多种药物耐药的结核病已成为HIV感染者中的主要问题。由于HIV感染与对结核病的易感性增高有关,或者使疾病的发展加速及死亡率增高,因此如HIV感染持续发展,则结核病的发病率将要进一步增加。所以迫切要求有新的方法,特别是免疫学方法来协助结核病的控制。
一、分子生物学方法在结核病诊断中的应用
(一)基因探针
DNA探针能有效地协助由临床标本培养物中分枝杆菌的快速鉴定和检出。可用放射性核素标级和非放射性核素标记的探针,如SNAP系统(Syngena,San Diego,CA),即使用辣根过氧化物酶(HRP)标记的DNA探针或用以丫叮酯标记的Accuprobe系统(Gen-Probe,San Diego,CA)。这些探针可用以鉴定在固体培养基上的分离菌落和BACTEC培养物中分枝杆菌属的直接鉴定。在Gen-Probe系统中多用于靶细菌核糖体RNA(rRNA)互补的单链DNA探针。Accuprobe系统在鉴定固体培养基上菌落中的结核分枝杆菌和鸟分枝杆菌-细胞内分枝杆菌复合体(MAI)时的敏感性,与放射性核素标记探针相同,但当其应用于鉴定BACTEC培养物中的结核分枝杆菌时,其敏感性就不及放射性核素标记探针。不过Accuprobe若与原始的BACTEC分离法合并使用,可使鉴定得以在3周内完成。这些探针使用方便,并能很快地获得结果,但它们所能检出的细菌种类有限,而且其特异性和敏感性问题尚未获解决。
核酸杂交检定法的敏感性可因应用分支的DNA信号扩增方法而大大地增加。此法系用双功能寡核苷酸探针,这种探针含有一种对靶分子特异的序列和另外一种能与第2种分枝寡核苷酸结合的序列。第2种寡核苷酸因而具有许多可以和耦联了酶类的第3种寡核苷酸相结合的结合部位。第3种寡核苷酸在有底物存在的情况下能产生可察见的信号。理论上这种方法能使杂交信号扩增10~100倍,可提高杂交检定法的检测限度少至每一标本中的100~1 000个细菌。
另一检测临床标本中结核分枝杆菌的信号扩增系统则是基于结核分枝杆菌23SrRNA的反向靶向捕捉。然后继之以具有复制酶的复制检察探针的扩增。复制酶检定法不因痰液的存在而被抑制。其结果可以完全与培养法所获得者相一致。
(二)定向扩增
细菌DN A或RN A特异区域扩增和检定技术的进展,促进了结核病实验室诊断质量的提高。应用于结核分枝杆菌实验室诊断的这类方法有:聚合酶链反应(PCR)扩增、链置换(SDA)扩增和连接酶链反应(LCR)扩增。
1.聚合酶链反应(PCR) 在已往的数年内曾有不少有关用各种基因元件作为靶模板对临床标本进行基于PCR的检验方法的报告(表10-1)。过去曾用以在PCR中检定编码r RN A或蛋白质的分枝杆菌基因序列的程系包括分子量65×103和38×103蛋白以及MPB64和MPB70或插入元件,如IS6110和IS986。其他系统则用重复序列或特异性限制性片段,如1.5×103 bp Eco R1-Bam H1。
表10-1 结核杆菌诊断中所用PCR检定法中的作用元件和引物

在PCR中应用最多的作用元件为插入元件IS6110,这种元件对结核分枝杆菌复合体细菌是特异的,而且一般有多个拷贝,因此较之单拷贝作用元件的敏感性为高。Eisenach等应用IS6110序列证实可用PCR由痰液标本诊断结核病。他们测试了162份痰液标本,其中有51份痰标本来自阳性直接涂片、阳性培养或两者均为阳性的病人。51份标本中有50份为PCR阳性。在68份来自非结核病或非分枝杆菌感染的病人的标本中,只有1份为假阳性。目前已有很多用PCR由临床标本中检查结核分枝杆菌的报告。
Nolte等用IS6110序列进行PCR,对313份痰液标本的结核分枝杆菌检查上与镜检和常规培养的结果进行比较。结果发现,PCR法能在110份涂片阳性标本中检出105份为PCR阳性(95%),而在14份涂片阴性标本中检出8份为PCR阳性(57%)。PCR法检查时未发现有假阳性(特异性100%)。Shawar等用相同的作用元件扩增,检出90%的涂片阳性标本和53%的涂片阴性标本。Clarridge等在对5 000多份痰液标本的相似研究中发现,PCR能在52病例的33例(63%)中快速地诊断涂片阴性,培养阳性病例,只有5.7%的标本为PCR阳性,培养阴性,致使其总的敏感性、特异性和阳性预期值(均系与培养法相比较)分别为83.5%、99%、94.2%。
虽然PCR能明显的提高诊断结核病的质量,但是它也有若干缺点有待解决。Walker等应用PCR扩增87例病人临床标本中的IS6110,结果发现在所有的6例活动期结核、18例过去曾患结核病人中的15例、9例与结核病人接触者中的5例以及54例患有与结核无关的肺部疾病病人中的9例的标本之中,都可鉴定出有IS6110DNA的存在。Yuen,Schulger以及Beige等都报告了相似的结果。阳性PCR结果虽是结核病存在的指标,但并非经常与有临床意义的疾病相关。
PCR检定法中尚存在一些技术上的问题。如在不同实验室所进行的PCR之间就存在着敏感性和特异性上很大的差异,因而需要有严格的质量控制。另外,如有能引起假阴性结果的PCR抑制物存在,以及在体内不同部位标本内只能分离出少量的DNA等,这些困难都需要在技术上有所改进。
2.PCR试剂盒
(1)Amplicor。虽然以往曾经研究过不少基于PCR扩增方法的简化步骤,但这些方法还是较为复杂,不适用于临床实验室内的常规应用。近年来所发展的一些商业化制品系统就可解决这一问题。这种系统之一为Roche诊断系统所发展的Amplicor检定法。这是一种用以扩增结核分枝杆菌16SrRNA内作用元件的PCR试剂盒。在6个不同的实验室内总共检验了2 173份标本的结果显示,Anplicor检定法的敏感性为86%。总共有95%的培养法证实的结核病可用Amplicor法得以诊断,而这些病人中只有72%可用直接镜检检出分枝杆菌。这一检定法的特异性为98%。Dilworth等比较了Amplicor PCR和IS6110PCR,发现两者的敏感性相等(96%),但Amplicor PCR的特异性(98%)较之IS5110PCR(79%)为高。
(2)AMTD。另一试剂盒为Gen-Probe所发展的扩增结核分枝杆菌直接试验(amplified mycobacterium tuberculosis direct test,AMTD)。这是一种直接由呼吸道标本直接鉴定结核分枝杆菌的检定法,并可在4~5h内完成。在此法中rRNA被扩增,扩增产物以特异性的化学发光探针检测。Vlaspolder等用AMTD由550份呼吸道和非呼吸道标本中诊断出340例结核病人,并与常轨培养法进行比较,他们发现敏感性为98.4%,特异性为98.9%;阳性和阴性预期值分别为93.8%和99.7%。
对这两试剂盒的应用也进行了比较,结果表明这两种核酸扩增方法在检测呼吸道标本中的结核杆菌时,都是很快速、敏感和特异的。
(3)链置换检定法(SDA)。SDA是一种体外的等温DNA扩增法。此法系用DNA聚合酶和限制性内切酶使能在2h内发生指数式扩增(约107倍)。种特异性SDA检定法是Becton和Dickinson研究中心所发展,用以检查结核分枝杆菌、鸟分枝杆菌或堪萨斯分枝杆菌。
(4)连接酶链反应(LCR)。LCR是Abbott实验室所发展的。Leckie等用这种方法检查痰液标本中的结核杆菌时具有很高的敏感性和特异性,而且在标本处理后5 h内能完成检查。
(三)PCR与物种形成
16S r RN A基因是在基因水平上扩增分枝杆菌核酸最为有用的作用元件,可以通过属特异性探针确定正确的扩增,或用一套物种探针或序列分析进行鉴别。每一分枝杆菌菌属均有一可供作为鉴定分枝杆菌菌属时所用的种特异性杂交探针的特殊可变区域,这些可变序列称为特征序列(signature sequence)。
PCR和地高辛配基(digoxigenin)标记属和种特异性寡核苷酸探针可用以检测属特异性寡核苷酸(作用元件16S r RN A)。
属特异性引物:5′TGCACACAGGCCACA AGGGA3′;
5′G A G T T TG A TCCT TGGCTCA GG A3′。
属特异探针:5′T T TCACGA ACA ACGCGACCA A3′。
种特异探针:5′A CCACA A G ACA T GCA TCCCG3′(结核分枝杆菌);
5′A CCAG A A GA CA T GCG TCT T G3′(鸟分枝杆菌)。
PCR的总反应量为100μl,其中含有2.5 U Taq聚合酶、50 mmol KCl、10 mmol Tris-H Cl(p H 8.3),1.5 mmol MgCl2,0.01%明胶、100 pmol两引物之中的一种和每种n N TP(d A TP,d CTP,d GTP,d T TP)各200μl。PCR条件为:93℃40循环周期1 min;60℃2 min;72℃6 min。
Kirschber等用作用于16S r RN A基因的分子检定法,在单一的扩增反应中可检出多种分枝杆菌病原体,其敏感性和特异性分别为84.5%和99.5%。这种方法操作方便,价廉,而且可在不用检测和鉴定分枝杆菌的单一菌种的多种扩增检定的情况下,即可鉴定所有的分枝杆菌菌种。
二、药物耐药性
异烟肼(IN H)、利福平和吡嗪酰胺(pyrazinamide)的联合应用,能有效地治疗结核杆菌药物敏感菌株感染。然而随着疾病病例的增加,对用以治疗的一种或多种一线抗结核药物的耐药结核杆菌分离菌株的耐药菌株数目日渐增多。多种耐药结核杆菌(MDR-TB)菌株(即至少是对IN H和利福平耐药的菌株)的出现可以引起许多国家内致死性流行暴发。有些M DRTB菌株甚至可对7种药物耐药。传统的测定药物敏感性的方法是检查药物对细菌生长的影响。然而进行常规药物敏感试验需要较长的时间,往往导致MDR-TB感染中的死亡后果。故在较短时间内得到正确分析结果是非常必要的。药物耐药性的增加、有效新型抗结核药物的缺乏以及对快速药敏试验的迫切需要,都要求进行有关研究,用以阐明药物耐药性的分子遗传基础。
(一)药物耐药性的分子基础
利福平是抗结核治疗中的主要药物。感染了利福平耐药菌株的病人,其预后较差,特别是尚有其他抗结核药物的耐药菌株存在时更为严重。利福平耐药性的原因是由于RN A聚合酶的改变。近年来对麻风分枝杆菌和结核分枝杆菌的整个RpoB基因都已进行了测序,而且在这两菌属中都已发现有与利福平耐药性有关的几种突变。在95%的利福平耐药结核杆菌分离菌株中,突变菌集中于RpoB基因的81bp区域内。
INH耐药性的分子基础尚不太清楚。Middlebrook早年观察到高度耐药的结核杆菌的触酶活性消失或大量降低。此种发现,再加上INH抗结核活性中与触酶过氧化物酶有关的生化作用,促使对编码触酶过氧化物酶的KatG基因进行克隆化和定性。早期研究报告在一高度INH耐药的结核杆菌菌株染色体KatG基因上有基因缺失,以后更为深入的研究证明,在耐药的分离菌株KatG基因上出现广泛的点突变和少量缺失或插入。另有一项不同的研究则是克隆能促使鸟分枝杆菌中对INH和乙硫异烟胺(ethionamide)耐药性增强的DNA片段,这一片段中发现含有INHA基因,此种基因编码与分枝酸的生物合成有关的酶,此酶也是活化药物的作用靶。KatG基因以及结核杆菌的34株耐药菌株和12株敏感菌株INHA位点上两基因的序列分析提示,约有84%的INH耐药菌株在这些区域内出现突变。相似的情况也出现于MDR-TB菌株。另有一种在INH耐药性中起作用的基因为编码烷基氢过氧化物酶的AhpC基因。
链霉素则是在核糖体的水平上起作用,阻止mRNA转译成为蛋白质。结核杆菌中高度的链霉素耐药性主要是由于编码核糖体蛋白S12的RpsL基因的错义突变(missense mutation)。链霉素耐药的临床分离菌株中最为常见的突变为密码子43处A至G碱基的转换,从而使赖氨酸为精氨酸所替代。少数菌株在由Rrs基因编码的16SrRNA的保守性环状结构上有突变发生。但在RpsL基因或约有30%链霉素耐药结核杆菌临床分离菌株的Rrs基因中则未发现有突变。
由于一线抗结核药物出现耐药性,促使人们对用环丙沙星(Ciprofloxacin)治疗结核感兴趣,这种药物的作用靶为DNA促旋酶(gyrase),此种酶与DNA的超螺旋和解旋有关,在转录和复制中很重要。DNA促旋酶由两个A亚单位和两个B亚单位所组成,分别由GyrA和GyrB基因编码。这些基因的突变可引起许多菌种对此类药物的高度耐药性。12株耐药结核杆菌株的分析发现在GyrA的4个残基中的一个残基上有点突变,其中半数影响残基94。在GyrB上则未发现突变。
(二)耐药分离菌株的鉴定
可用核苷酸测序或检查点突变的技术来鉴定突变。Telenti等用PCR和单链构象多态性(SSCP)检测RpoB基因上的突变。PCR中应用两个引物,即5′TGCACGTCGCGGACCTCCA3′和5′TCGCCGCGATCAAGGAGT3′,在有32P-dCTP存在的情况下生成一放射性标记的157bp扩增的产物。扩增的其他试剂为:50μl的反应容积中含有50mmol KCl、10mmol Tris-HCl(pH 8.3)、1.5mmol MgCl2、10%甘油、每种dNTP各200μl,每种引物0.5μl及1.25UTaq聚合酶。所用的循环参数为:40周期的94℃1min变性;55℃1min退火;72℃1min延伸,最后继之以72℃10min的延伸。稀释的变性PCR产物在变性测序形式凝胶中进行电泳,并且进行放射自显影。应用PCR-SSCP可在66株利福平耐药的结核杆菌分离菌株中鉴定出84株,这种方法是一种很有希望的确定耐药行出现的技术,在检测利福平耐药性上甚为有效(成功率为97%),这点之所以重要,是因为利福平耐药性是预测其他药物耐受性有用的代理指标。PCR-SSCP在检测IN H耐药性上并不适用,因为此时涉及多种耐药机制,故需用多个试验进行检测。
另外尚发展了一种检测RpoB基因上点突变的简化方法(Inno-Lipa Rif TB)(Innogenetics,Beigium)。在此方法中,结核分枝杆菌RpoB基因的250 bp片段用PCR扩增,试剂盒中的引物为:5′GGTCGGCA TGTCGCGGA TGG3′和5′GCACGTCGCGGACCTCCAGC3′,两引物均在5′端生物素酰化。所用扩增条件:95℃5 min变性,继之以95℃1 min;55℃1 min;72℃1 min共30个循环及最后72℃10 min延伸,并与固定于条带膜的5个野生型和4个突变型的寡核苷酸探针杂交,用比比色反应确定是否有结合发生。根据其结果即可鉴定耐药菌株(表10-2)。
表10-2 Inno-Lipa法中所用探针(野生型和突变型)的基因序列

测定药物对细菌生长或细菌代谢影响的方法可以不必对各种不同的药物设计多种试验进行检测。Kawa等用结核分枝杆菌快速诊断系统(Gen-Probe,San Diego,CA)检测以IN H治疗的结核杆菌培养物中的r RN A水平。他们能在3~5 d内鉴别IN H耐药和IN H敏感菌株,而常规的培养法则需要21~28 d之久。
分子遗传学方法用以检测结核分枝杆菌耐药性的另一应用是基于报道基因噬菌体系统,即将编码荧光素酶的基因(lux)插入分枝杆菌噬菌体,使其当此种噬菌体感染活的结核分枝杆菌时产生化学发光信号。细菌培养物与试验药物短期孵育后与报道基因噬菌体混合。耐药活菌的噬菌体感染可引起化学发光,而敏感菌株则不能发光。此种检定法能在细菌生长培养的18~24 h内获得药敏试验的结果。但所需菌量较多(106个细菌),故尚未用于临床标本的检查。
三、流行病学研究
控制结核病的一项重要因素为鉴定疾病流行和追踪特定结核分枝杆菌传播的能力。感染个体中菌株的分型在追踪传染来源上起着重要的作用。过去对于结核病传播的证实是根据出现暴发性流行的聚集、具有特殊耐药型程式的传播以及接触活动期病人后结核菌素皮肤试验的转阳率。分离菌株对抗生素敏感性程式的比较可用以追踪不常见耐药性的扩散,但是可能的不同敏感性程式的数目不多,以及分枝杆菌菌种当其接触了抗结核药物后,耐药性程式可以发生改变,故其应用是很有限的。噬菌体分型虽可用以鉴别菌株,但在结核病的流行病学研究的应用价值上也很有限,因为可供使用的各种噬菌体型别不多。血清学分型在鉴别结核分枝杆菌菌株上是不成功的。同样,质粒分型也不适用于结核杆菌,因为质粒为数不多或根本就无质粒。另外一些分枝杆菌分型方法,如细胞蛋白质的电泳分析和生化异质性也存在有重复性和各类型别不多的问题。多位点酶电泳法(MLEE)曾用以鉴别分枝杆菌,但这种方法却不能区别结核分枝杆菌和牛分枝杆菌。
可用能识别DNA分子中特异的核苷酸序列限制性内切酶来检定密切相关菌株间的相互关系。Imeda等报告各种限制性内切酶所产生DNA片段的分布程式在结核分枝杆菌的代表菌株中是难以区分的。而Collins等用此方法成功地区别牛结核菌株,但染色体DNA总的限制性程式不易分析。分析限制性片段异质性较为敏感的技术是用由克隆的DNA片段组成的分子探针与之杂交。Eisenach等用此方法证实,经过选择的DNA探针在测定结核分枝杆菌菌株间的差异甚为有用。
近年应用重复性元件的研究显示了结核分枝杆菌染色体上的多态性。有两类重复性DNA元件可用于结核病的分子流行病学。插入序列(IS)元件具有能在基因组上移动的能力,其大小约为1 300bp,重复性DNA序列大小在3~36bp之间。与插入元件有关的DNA多态性可能是由于其能移动至基因组上的随机位置所致。只是对存在于多数MTB菌株中的同向重复(DR)区域中由小型重复元件引起基因重排的性质进行了研究,DR位点含有多个保守性的36bp大小的DR,其间隔有非重复性、长度为34~41bp的间隔序列。菌株在DR的数目和是否存在有特殊的间隔序列上有所不同。很有可能同样的机制亦可见于与其他小型重复序列有关的DNA多态性,如多态性富含GC重复序列(PGR)、主要多态性串联重复体(MPTR)和GTG重复体。
(一)IS6110限制性片段长度多态性(RFLP)
插入元件IS6110是结核病流行病学研究中最常用的标记之一,这是因为利用这种元件可以获得高度的鉴别能力,这一方法的优点是:结核分枝杆菌菌株带有IS6110的多个拷贝,而且IS5110元件在结核分枝杆菌基因组中的确切位置在菌株与菌株之间各不相同。故每一结核杆菌菌株都可有其独特的DNA指纹。IS6110RFLP技术的要点为,由培养的细菌提取整个基因组DNA,再继之以用限制性内切酶PvuII的消化,所形成的DNA片段以凝胶电泳分离,转移至尼龙膜,并与针对插入元件IS6110的标记探针进行杂交。这种技术已被证实为鉴别结核杆菌菌株的一种可靠及可重复的方法。分离菌株的指纹模式在实验室保存和动物传代中都很稳定。而且在慢性结核病人的长期过程中很少或无变化。基于IS6110的DNA指纹在耐药性发生过程中也无改变。
IS6110-RFLP法已广泛应用于结核病的流行病学研究,如流行暴发、社团中的传播和多种药物耐药菌株的播散,也可用于检测可疑的实验室内交叉污染。这一方法尚可用以区别新近获得的感染和复发。IS6110-RFLP法已用于确定特定结核分枝杆菌菌株的感染性是否可以受到宿主的HIV感染状况的影响。结果发现,HIV阳性和HIV阴性个体对特定结核分枝杆菌感染具有同等的危险性。
应用IS6110-RFLP法也进行了有关人种间结核病感染的研究,结果显示,即使有适当的结核控制基础结构存在,也只有一部分结核病的主要传播途径可以通过常规的接触追踪方式发现。根据如有相同的分离菌株存在则是新近传播指标的推论,美国旧金山和纽约所进行的两项研究证实:新近获得的感染,而不是隐性疾病的再活化,在结核病发病率增高上起作用。
IS6110-RFLP程式表现地区性变化。一般而言,结核分枝杆菌高度流行的国家,如中非共和国较之低度流行的国家,如荷兰等国,其DN A多态性不多。在中国所进行的结核分枝杆菌菌株人群结构分析证实,多数菌株均属于遗传密切相关的群体。
IS6110-RFLP分型法也有若干限制。其中的一个主要缺点为操作步骤较繁,需时较长。由于这一方法需要有大量提取的DN A,初次培养物必须有大量的生长,或须进行再次培养以获得足够的细菌。因此,整个RFLP过程常须3~4周方可获得可供判断的结果。此种方法的另一限制是在亚洲发现有无IS6110拷贝的结核分枝杆菌菌株。
寡核苷酸(GTG)5亦可用以分析结核分枝杆菌DN A的多态性,特别是用于只有少数或无IS6110序列拷贝的分离菌株的分型。
(二)脉冲凝胶电泳(PFGE)
RFLP的一种修正技术为PFGE。这一技术亦可用于结核分枝杆菌分离菌株的分型。此方法中所用的限制性内切酶可将基因组DN A割切成少数极大的片段。Zhang等应用PFGE可将26株结核分枝杆菌分离菌株区分为不同的型别,而且与IS6110-RFLP结果一致。如同IS6110-RFLP一样,这一技术也需要基因组DN A,因而也需要培养细菌。
(三)基于PCR的分型
为了克服需要培养细菌的困难,不同的实验室利用各种结核分枝杆菌DN A作用元件发展了许多基于PCR的分型方法(表10-3)。Palittapongarnpim等用任意引物(A P)PCR鉴别结核分枝杆菌分离菌株,他们证实了不同菌株中的多态性,而且鉴别的程度与在IS5110-RFLP中所获得者基本上相同。随机扩增多态性DN A分析法(RAPD)则是一种快速而简易的指纹分析方法,已成功地应用于结核分枝杆菌菌株的鉴定。但要获得可靠的结果,必须使用大量的引物。Abed等利用16~23S r RN A间隔区作为RAPD的模板DN A,用任意引物即能有效地鉴别结核分枝杆菌菌株。他们证实,在此种条件下所进行的RAPD较之以整个基因组DN A作为作用元件时更为有效。
表10-3 用于鉴定结核分枝杆菌复合体分离菌株的各种基于PCR分型方法中的引物
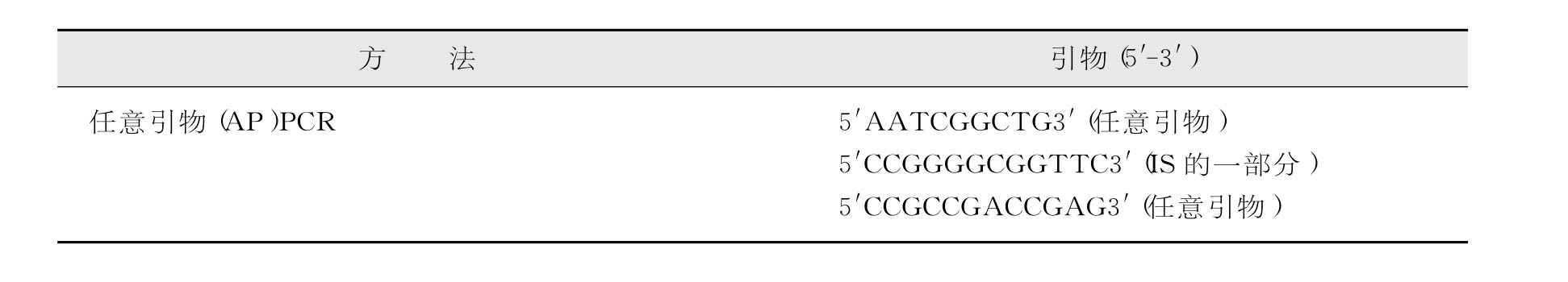
(续 表)

混合接头PCR利用连接于限制性片段上的寡核苷酸接头和IS6110作为引物作用元件对鉴别性扩增子进行PCR介导的扩增。Ross等用结合于IS6110末端的引物扩增这一元件的密切间隔的拷贝。Plikaytis等将MPTR和IS6110合并使用,成为较之一般的指纹分析技术更为方便的IS6110扩增指纹分型法。另外有人利用存在于INH耐药性基因(KatG)上游的可变性序列作为基于PCR的结核分枝杆菌分型方法。
另外一种新的亚型分型法则是利用位于重复元件IS6110和PGR之间的两个拷贝的结核杆菌DNA片段的扩增。这种方法的合理性是根据这样的事实,即重复元件间的距离和IS6110和PGR的拷贝数在菌株之间各不相同。这种方法称为双种重复元件PCR(DREPCR),能将46种临床分离菌株区分为25个不同的型别。
四、总结
DN A扩增法将为21世纪中提供进一步缩短诊断结核和进行结核分枝杆菌药敏试验的时间。可以由数周缩短至数日,甚至数小时。快速筛选可能的MDR-TB病例将成为其主要的应用。IS6110-RFLP指纹分析有助于对于结核病传播的了解,在某些情况下,如在HIV病房中的医院内传播,很有需要有由临床标本内直接对结核分枝杆菌分离菌株进行分型的快速试验,而不须要借助于培养方法。目前已有很多基于DN A扩增的方法,但是这些方法都需要进一步修改和简化,使之成为可在结核分枝杆菌实验室中应用的方法。这些快速试验的应用必将有助于将来对于结核病复活的控制。表10-4中总结了结核分枝杆菌感染的诊断、药物耐受性以及结核分枝杆菌物种形成和菌株分型中所用的常用、改良和快速分子生物学方法。
表10-4 结核分枝杆菌的诊断、药物敏感性和分型所用方法的总结

大多数的结核病病例发现于经济不发达地区,在这些地区中。一些所谓“高科技”和价高的现代DN A扩增技术并不能完全替代传统的涂片检查方法。必须在世界上因结核病而夺去最多人群生命的地区内,进一步进行有关结核病的控制中能常规应用方法的研究。
(余传霖)
参考文献
1.Drake T A,Hingler JA,Berlin GGW,et al.Rapid identification of Mycobacterium avium complex in culture using DN A probes.J Clin Microbiol,1987,25:1 442~1 445
2.Lim SD,Todd J,Loper J,et al.Genotypic identification of pathogenic mycobacterial species by using a non-rodioactive oligonucleotide probe.J Clin Microbio,1991,29:1 276~1 278
3.Goto M,Oka S,Okuzumi K,et al.Evaluation of acridinum ester labelled DNA probes for identification of Mycobacterium tuberculosis and Mycobacterium intracellulare complex in culyutr.J Clin Microbiol,1991,29:2 473~2 476
4.Eisenach KD,Cave MD,Bates JH,et al.Polumerase chain reaction amplification of a repetitive DNA sequence for Mycobacterium tuberculosis.J Infect Dis,1990,161:977~981
5.Walker GT,Nedeau JG,Spears PA,et al.Multtiplex strand displacement amplification(SDA)and detection of DNA sequences from Mycobacterium tuberculosis and other mycobacteria.Nucleic Acids Res,1994,22:2 670~2 677
6.Leckie G,Gao J,Davis A,et al.Ligase chain reaction(LCR)amplification for direct detection of Mycobacterium tuberculosis in clinical specimens in Abstracts of the 94th General Meeting of the American Society for Microbiologists.Las Vegas,Abst-U 96American Society for Microbiology Washington DC,1994
7.Anderson AB,Thybo S,GODfrey-Faussett P,et al.Polymerase chain reaction for detection of Mycobacterium tuberculosis in sputum.Eur J Clin Microbiol,1993,12:922~927
8.Cousins DV,Wilson SD,Francis BR,et al.Use of polymerase chain reaction for rapid diagnosis of tuberculosis.J Clin Microbiol.1992,30:255~258
9.Hermans PWM,van Soolingen D,Dale JW,et al.Insertion element IS986from Mycobacterium tuberculosis:a useful tool for diagnosis and epidemiology of tuberculosis.J Clin Microbiol,1990,28:2 051~2 058
10.De Wit D,Wootton M,Allan B,et al.Simple method for production of internal control DNA for Mycobacterium tuberculosis polymerase chain reaction.Am Rev Respir Dis,1993,144:1 160~1 163
11.Thierry D,Cave MD,Eisenach KD,et al.IS6110,an IS-like element of Mycobacterium tuberculosis complex.Nucleic Acids Res,1990,18:188
12.Eisenach KD,Sifford MD,Cave MD,et al.Detection of Mycobacterium tuberculosis in sputum samples using polymerase chain reaction.Am Rev Respr Dis,1991,144:1 160~1 163
13.Nolte FS,Metchell B,McGowen JE Jr,et al.Direct detection of Mycobacterium tuberculosis in sputum by polymerase chain reaction and DNA hybridization.J Clin Microbiol,1993,31:1 777~1 782
14.Shawar RM,El-Zaatari FAK,Nataraj A,et al.Detection of Mycobacterium tuberculosis and nonisotopic hybridization methods.J Clin Microbiol,1993,31:61~65
15.Clarridge JER,Shawar RM,Shinnick TM,et al.Large scale use of polymerase chain reaction for detection of Mycobacterium tuberculosis in a routine mycobacteriology laboratory.J Clin Microbiol,1993,31:2 049~2 056
16.Walker DA,Taylor IK,Mitchell DM,et al.Comparison of polymerase chain reaction amplification of two mycobacterial DNA sequences IS6110and the 65-kDa antigen gene,in the diagnosis of tuberculosis.Thorax,1992,47:690~694
17.Yuen LK,Ross BC,Jackson KM,et al.Characterization of Mycobacterium tuberculosis strains from Vietnamese patients by southern blot hybridization.J Clin Microbiol,1993,31:1 615~1 618
18.Schulger NW,Kinney D,Harkin TJ,et al.Clinical utility of the polymerase chain reaction in the diagnosis of infections due to Mycobacterium tuberculosis.Chest,1994,105:1 116~1 121
19.Beige J,Lokies J,Schaberg T,et al.Clinical evaluation of Mycobacterium tuberculosis assay.J Clin Microbiol,1995,33:90~95
20.Noordhoek GT,Kolk AHJ,Bjune G,et al.Sensitivity and specificity of PCR for detection of Mycobacteri-
um tuberculosis:a blind comparison study among seven laboratories.J Clin microbiol,1994,32:177~284
21.Kolk A HJ,Noordhoek GT,De Leeuw O,et al.Mycobacterium smegmatis strain for detection of Mycobacterium tuberculosis by PCR used as internal control for inhibition of amplification and quantification of bacteria.J Clin Microbiol,1994,32:1 354~1 356
22.Victor T,du Toit R,van Helden PD.Purification of sputum samples through sucrose improves detection of Mycobacterium tuberculosis by polymerase chain reaction.J Clin Microbiol,1992,30:1 514~1 517
23.Buck GE,O’Hara LC,Summergill JT.Rapid,simple method for treating clinical specimens containing Mycobacterium tuberculosis to remove DNA for polymerase chain reaction.J Clin Microbiol,1992,30:1 331~1 334
24.Dilworth JP,Goyal M,Young DB,et al.Comparison of polymerase chain reaction for IS6110 and amplicor in the diagnosis of tuberculosis.Thorax,1996,51:320~322
25.Vlaspolder F,Singer P,Roggeveen C.Diagnosis value of an amplification method(Gen-Probe)compared with that of culture for diagnosis of tuberculosis.J Clin Micrtobiol,1995,33:2 699~2 703
26.Kirschner P,Rosenau J,Springer B,et al.Diagnosis of mycobacterial infections by nucleic acid amplification:18 month prospective study.J Clin Microbiol,1996,34:304~312
27.Musser JM.Antimicrobial agent resistance in mycobacteria:molecular genetic insights.Clin Microbiol Rev,1995,8:496~514
28.Miller LP,Crawford JT,Shinnick T M.The rpoB gene of Mycobacterium tuberculosis.Antimicrob Agents Cgemother,1994,38:313~319
29.Telenti A,Imboden P,Marchesi F,et al.Detection of rifampicin resistance mutations in Mycobacterium tuberculosis.Lancet,1993,341:805~811
30.Heym B.Zhang Y,Poulet S,et al.Characterization of the kat G gene encoding a catalase-peroxidase required for the isoniaazid susceptibility for Mycobacterium tuberculosis.J Bacterol,1993,175:4 255~4 259
31.Altamirano M,Marostenmaki J,Wong A,et al.Mutations in the catalase-peroxidase gene from isoniazidresistant Mycibacterium tuberculosis isolates.J Infect Dis,1994,169:1 162~1 165
32.Banerjee A,Dubnau F,Quenmard A,et al.Inh A,a gene encoding a target for isoniazid and ethionamide in Mycobacterium tuberculosis.Science,1994,263:227~230
33.Heym B,Honore N,Truffot-Pernot C,et al.Implications of multidrug resistance for the future of short course chemotherapy of tuberculosis:a molecular study.Lancet,1994,30:277~279
34.Wilson T M,Collins DM.AhpC,a gene involved in isoniazid resistance of the Mycobacterium tuberculosis complex.Mol Microbiol,1996,19:1 025~1 034
35.Nair J,Rouse DA,Bai G H,et al.The rps L gene and streptomycin resistance in simple and multiple drugresistant strains of Mycobacterium tuberculosis.Mol Microbiol,1993,10:521~527
36.Finken M,Kirschner P,Meier A,et al.Molecular basis of streptomycin-resistance in Mycobacterium tuberculosis:alterations of the ribosomal protein S12 gwne and point mutations within a functional 16S ribosomal RN A pseudoknot.Mol Microbiol,1993,9:1 239~1 246
37.Takiff H E,Cimino M,Musso NC,et al.Efflux pump of the protein antiporter family confers low level fluoroquinolone resistance in Mycobacterium smegmatis.Proc Natl Acad Sci USA,1996,93:362~366
38.Jacobs WR,Jr.Barletta RG,Udani R,et al.Rapid assesment of drug susceptibility of Mycobacterium tuberculosis by means of luciferase reporter phages.Science,1993,260:810~822
39.Plikaytis BB,Marden JL,Crawford JT,et al.Multiplex PCR assay specific for the multidrug resistant strain W of Mycobacterium tuberculosis.J Clin Microbiol,1994,32:1 542~1 546
40.van Soolingen D,Hermans PWM,de HaasPEW,et al.Occurrence and stability of insertion sequences in Mycobacterium tuberculosis complex strains:evaluation of an insertion sequence dependent DNA polymorphism as a tool in the epidemiology of tuberculosis.J Clin Microbiol,1991,29:2 578~2 586
41.Daley CL,Small PM,Scheeter GF,et al.An outbreak of tuberculosis with accelerated progression among persons infected with the human immunodeficiency virus:an analysis using restriction fragment length polymorphism.New Engl J Med,1992,326:231~235
42.Small PM,Shafer RW,Hopewell PC,et al.Exogenous reinfection with multidrug resistant Mycobacterium tuberculosis in patients with advanced HIV infection.New Engl J Med,1993,328:1 137~1 144
43.Yang ZH,Mtoni I,Chonde M,et al.DNA fingerprinting and phenotyping of Mycobacterium tuberculosis isolates from human immunodeficiency virus(HIV)-seropositive and HIV seronegative patients in Tanzania.J Clin Microbiol,1995,33:1 064~1 069
44.Small PM,Hopewell PC,Singh SP,et al.The epidemiology of tuberculosis in San Francisco:apopulation based study using conventional and molecular methods.New Engl J Med,1994,330:1 703~1 709
45.Genewin A,Telenti A,Bernasconi C,et al.Molecular approach to identify route of transmission of tuberculosis in the community.Lancet,1993,342:841~644
46.van Soolingen D,Qian L,de Haas PEW,et al.Predominance of a single genotype of Mycobacterium tuberculosis in countries of East Asia.J Clin Microbiol,1995,33:3 234~3 238
47.Zhang Y,Mazurrrrek GH,Cave MD,et al.DNA polymorphism in strains of Mycobacterium tuberculosis analyzed by pulsed field gel electrophoresis:a tool for epidemiology.J Clin Microbiol,1992,30:1 551~1 556
48.Palittapongarnpim P,Chomye S,Fanning A,et al.DNA fingerprinting of Mycobacterium tuberculosis isolates byligation mediated polymerase chain reaction.Nucleic Acids Res,1993,21:761~762
49.Linton CJ,Jalal Hm,Leeming JP,et al.Rapid discrimination of Mycobacterium tuberculosis strains by random amplified polymorphic DNA analysis.J Clin Microbiol,1994,32:2 169~2 174
50.Abed Y,Davin-Regli A,Bollet C,et al.Efficient discrimination of Mycobacterium tuberculosis strains by 16S~23Sspacer region based random amplified polymorphic DNA analysis.J Clin Microbiol,1995,33:1 418~1 420
51.Haas WH,Butler WR,Woodley CL,et al.Mixed linker polymerase chain reaction:a new method for rapid fingerprinting of isolates of Mycobacterium tuberculosis complex.J Clin Microbiol,1993,31:1 293~1 298
52.Ross BC,Dwyer B.Rapid,simple method for typing isolates of Mycobacterium tuberculosis by using the polymerase chain reaction.J Clin Microbiol,1993,31:329~334
53.Goyal M,Young D,Zhang Y,et al.PCR amplification of variable sequence upstream of katG gene to subdivide strains of Mycobacterium tuberculosis complex.J Clin Microbiol,1994,32:3 070~3 071
54.Pilkaytis BB,Crawford JT,Woodley CL,et al.Rapid amplification based fingerprinting of Mycobacterium tuberculosis.J Gen Microbiol,1993,139:1 537~1 542
55.Friedman CR,Stoeckle MY,Johnson WD,et al.Double-repetitive-element PCR method for subtyping Mycobacterium tuberculosis clinical isolates.J Clin Microbiol,1995,33:1 383~1 384
免责声明:以上内容源自网络,版权归原作者所有,如有侵犯您的原创版权请告知,我们将尽快删除相关内容。

















