微卫星是真核生物基因组中广泛分布的简单重复DNA片段,一般由1~6个核苷酸为基本单位组成。与其他分子标记相比,微卫星标记具有多态性丰富、遵循孟德尔分离定律、共显性遗传、易于PCR 扩增、条带易于识别等特点,因此,广泛应用于群体遗传学分析、系谱认证、QTL定位、遗传图谱的构建及种质鉴定等多方面的研究(胡则辉等,2006)。本研究采用富集文库—菌落原位杂交法对三疣梭子蟹微卫星位点进行筛选,开发了微卫星标记,并估算了这些位点的主要遗传学参数,为后继大量开发三疣梭子蟹微卫星标记提供了一种可行性方法,同时为开展三疣梭子蟹群体遗传多样性分析、遗传连锁图谱的构建和QTL定位奠定了基础。
2.1.1 微卫星富集文库的构建
实验所用样品是于2005年取自辽宁省鸭绿江口附近海域的野生三疣梭子蟹,共30只,取其大螯,280 ℃保存。提取三疣梭子蟹大螯部肌肉基因组DNA,采用限制性内切酶Sau3AI进行酶切反应,回收400~1.5.0 bp左右的片段,与接头进行连接,经杂交与洗膜、TA克隆及转化,菌落重排后,进行转膜及洗膜,获得阳性信号后挑选阳性克隆,进行测序。
通过对基因组文库DNA克隆随机测序,共获得了4164个DNA随机克隆源序列。利用软件进行序列处理和拼接,最终得到的709个DNA克隆,每个序列的长度从500~1.5.0 bp不等,代表着622409个碱基的基因组总长度。
对拼装后的序列进行分析,查找微卫星序列。共找到了827个重复序列,微卫星重复序列(1~6 bp重复)为697个,占重复序列总数目的比例为84.28%。统计微卫星重复类型,以两碱基重复数目最多为445个,占微卫星序列总数目的63.84%(图1)。
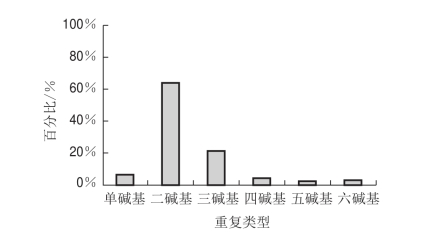
图1 不同重复类型的微卫星序列数目及其百分比
在同类型的重复序列中,各重复拷贝类别占的比例也各不相同。在46个单碱基重复类型中,重复拷贝类别全部为A型,没有发现核心序列为C型的重复拷贝类别。两碱基中,AG重复拷贝类别最多,为214个,占两碱基总重复序列数目的48.09%;其次是AC和AT,各为187(42.02%)和43(9.66%)个。只发现1个核心序列为GC的重复拷贝类别,即(GC)14,占两碱基总重复序列数目的0.23%。三碱基重复中,共发现8种重复拷贝类别(表2)。
表2 1-3碱基重复类型中重复拷贝类别及其在所属重复类型中的百分比
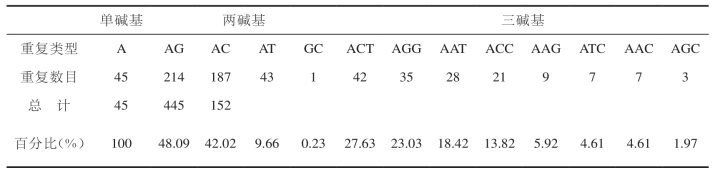
四碱基重复类型中,AGAC重复拷贝类别最多,共14个;五碱基重复类型中,AACCT重复拷贝类别最多,共6个;六碱基重复类型中,AGGGGA重复拷贝类别最多,共3个。其他分别是:CTCTCC共2个;TCTTCC共2个;TCCTCG共1个;AAAAGA 共1个;TACTGC共1个。使用引物设计软件在其核心序列的两端设计相应的PCR引物,共合成微卫星引物52对。选择了27对引物进行合成,引物的核心序列已经在GenBank进行了注册,登录序列号分别为EU26.6.0 ~ EU267706(表2)。
2.1.2 部分基因组文库SSR的筛选与评价
2.1.2.1 菌落原位法筛选的多态性SSRs群体评价
利用菌落原位杂交法,采用(AC)15和(AG)15两种探针,共筛选得到269个克隆。从中任意选取150个克隆进行测序,得到124个克隆的序列,106个含重复序列的克隆中全部只含有微卫星位点(100%),阳性克隆率为85.48%。利用软件进行序列拼接和识别,得到103个独立克隆序列,共176个微卫星位点。GenBank注册号为GQ466018-GQ466043和GU177130-GU177207。(105个)

图2 微卫星位点Pot54在三疣梭子蟹30个个体中扩增得到的聚丙烯酰胺凝胶电泳图
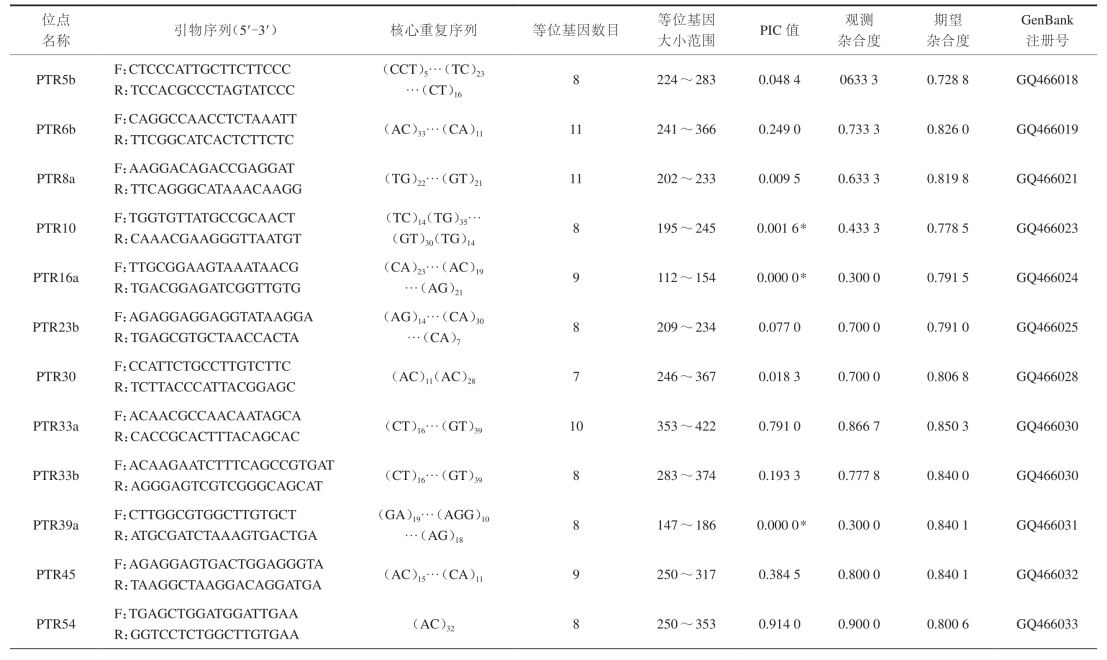
应用30个野生三疣梭子蟹个体对30个位点进行多态性评价,结果显示几乎每个实验个体在各个微卫星位点上都产生了较为清晰的扩增条带,其中30个(53.6%)位点具有多态性。读取全部位点在所有个体中的基因型,30个位点共获得了238个等位基因,平均每个多态性位点扩增得到8.0个等位基因。每个微卫星位点的等位基因数、核心序列、Tm值、PIC值、Ho、He、GenBank注册号见表3。
表3 三疣梭子蟹30个微卫星标记的基本特征及主要遗传学数据的估算
续表

续表
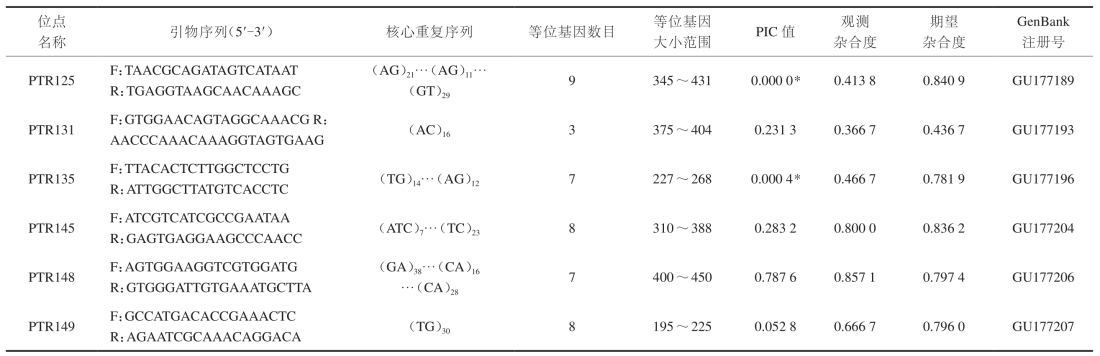
注:*表示严重偏离哈代-温伯格平衡的位点
不同的位点获得的等位基因数差异较大,从3~13个不等,呈现较高的多态性。获得的30个多态性基因座位中有8个座位的观测杂合度和期望杂合度有显著的差异;哈代—温伯格平衡检验发现,这些位点的偏离是由于杂合子的严重缺乏(P , 0.005表中*所示)造成的。相关分析指出杂合子缺失大多是由于无效等位基因引起的。微卫星在进行PCR扩增时偶尔会出现无效等位基因(null allele)的现象,即无特异性的扩增产物。这可能是微卫星引物的结合部位的点突变、插入或缺失阻碍微卫星的扩增,而且这种突变并没有在群体中被固定,则只有一部分等位基因没有被扩增出来,因此在电泳时,实际为杂合的位点,仅出现单条电泳带而呈现纯合现象,另一部分个体则可能由于含有两个无效等位基因而得不到相应的扩增产物。所以无效等位基因如果不被识别出来,则会导致群体中纯合子过剩或杂合子缺失的现象,从而出现观测杂合度与期望杂合度偏离的现象。
2.1.2.2 FIASCO菌落原位法筛选的多态性SSRs群体评价
利用Primer Premier5.0根据测序所得微卫星序列设计了105对微卫星引物,其中81对引物能够得到清晰的预期长度的扩增片段,其中56对引物表现出多态性(图3)。

图3 部分引物多态性筛选结果
利用FIASCO方法构建的三疣梭子蟹微卫星富集文库,对含微卫星的DNA序列设计了71对引物并对其进行了多态性筛选,筛选出21对多态的微卫星引物,已在GenBank注册。
利用微卫星引物对三疣梭子蟹海州湾群体30个个体进行遗传多样性分析。对每一个微卫星位点的等位基因数目(其中纯合个体的等位基因按两次计算)进行统计。根据各个微卫星位点各种基因型频率计算其观察杂合度,根据各个微卫星位点的等位基因频率,分别计算它们的期望杂合度和多态信息含量(PIC)。在三疣梭子蟹30个样本的21个微卫星位点中,共获得了188个等位基因,平均每个位点扩增得到8.9个等位基因。不同引物获得的等位基因数差异较大,从3~13个不等,其中Pot8、Pot37、Pot48、Pot53、Pot54、Pot66等6个位点分别获得了11、12、12、11、13、11个等位基因,而Pot46只获得了3个等位基因,等位基因的大小为131~312 bp,基本符合引物设计时理论产物长度。期望杂合度的范围从0.716到0.913,表明它们都有较高的杂合度。PIC值为0.6.9 ~ 0.889,21个位点的PIC值高于0.5,表明这些微卫星位点在三疣梭子蟹中均具有较高的信息含量。位点Pot66的观察杂合度与期望值有较大的出入,其他微卫星位点的这两值均基本相符。
2.1.3 不同地理群体三疣梭子蟹的遗传结构分析
从筛选引物中根据遗传学信息评价,选择多态性适中、扩增产物稳定、条带清晰的标记选取8对引物(PTR33a,PTR45,PTR93,PTR95,PTR98a,PTR103b,PTR112,PTR145)。选取我国由南到北5个不同地理区域的三疣梭子蟹:舟山野生群体(ZS)采自浙江舟山群岛近海、海州湾野生群体(HZ)采自江苏连云港市海州湾近海、莱州湾野生群体(LZ)采自山东省昌邑市下营港近海、鸭绿江口野生群体(YL)采自辽宁省鸭绿江口近海,会场野生群体(HC)采自青岛即墨会场,每个群体24只,用于三疣梭子蟹的遗传多样性分析。
2.1.3.1 8个位点在群体中的扩增结果
8个位点在五个群体中都扩增出清晰稳定的带谱,如图4为位点PTR33a在三疣梭子蟹5个群体中的扩增情况。
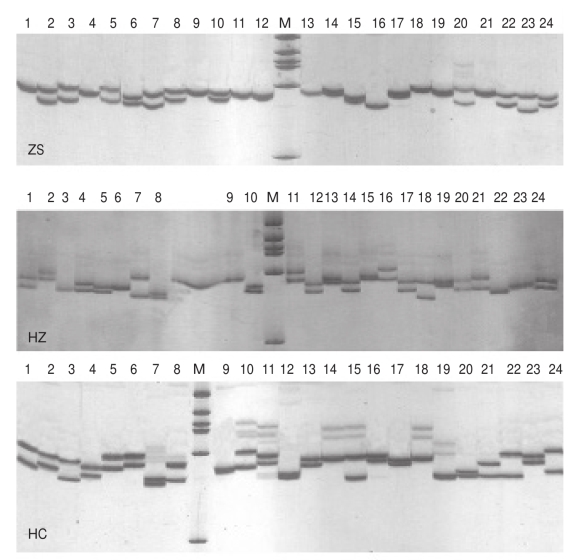
图4 位点PTR33a在三疣梭子蟹5个群体中的扩增情况(Ⅰ)

图4 位点PTR33a在三疣梭子蟹5个群体中的扩增情况(Ⅱ)
(M由上至下分别为267 bp,234 bp,213 bp,192 bp,184 bp,124 bp)
2.1.3.2 等位基因数和有效等位基因数
8个位点在5个三疣梭子蟹群体的120个个体中共扩增得到72个等位基因,不同引物获得的等位基因数从6~12不等,平均每个位点获得的等位基因数目为9.0个,其中位点PTR112获得了12个等位基因,等位基因数最多;PTR33a获得的等位基因数次之,为11个;位点PTR95和PTR103b都获得了6个等位基因。有效等位基因数在2.905.8 ~ 7.7.0 1之间,平均每个位点检测的有效等位基因数目为5.4677个,如表4所示。
2.1.3.3 多态信息含量
多态信息含量是衡量微卫星位点变异程度高低的一个标准,当PIC . 0.5时,该位点为高度多态性位点;当0.25 , PIC , 0.5时,为中度多态性位点;当PIC , 0.25时,为低度多态性位点。8个位点在5个群体中得到的多态信息含量有所不同,其中位点PTR112在HZ群体中获得的多态信息含量最高为0.8.4 9,而位点PTR103b在HZ群体中的多态信息含量最低为0.3.9 5。统计各位点在各群体中的PIC值发现,只有两个PIC值低于0.5(位点PTR103b在HZ群体和位点PTR95在ZS群体),属于中度多态性位点,其他PIC值均大于0.5。从8个位点在每个群体中的平均多态信息含量来看,PIC值从0.674.9 ~ 0.7.3 9不等,均大于0.5;从各微卫星位点的平均多态信息含量来看,PIC值在0.539.4 ~ 0.8.0 3之间,同样都大于0.5,平均多态信息含量为0.6.6 7。由此可见,所选择的微卫星位点具有丰富的多态性,能够在分子水平上很好地反映5个地理群体间和群体内的遗传关系。
表4 三疣梭子蟹5个群体8个微卫星座位的等位基因数、有效等位基因数、多态信息含量、各个群体观测杂合度、期望杂合度
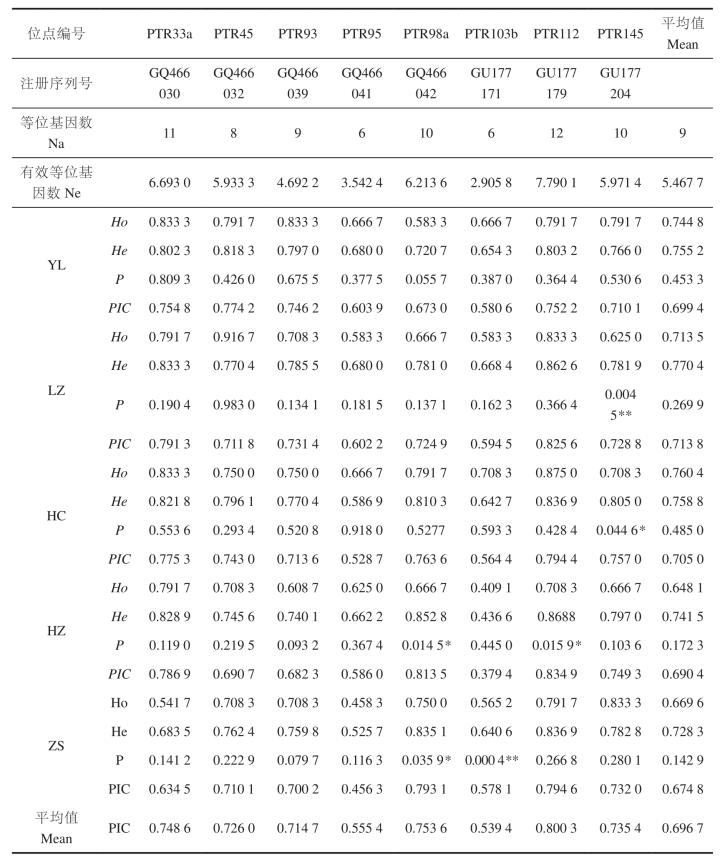
注:*P , 0.05显著;** P , 0.01极显著
2.1.3.4 观测杂合度、期望杂合度及Hardy-Weinberg平衡检验
由表5可知,8个位点在5个群体中所反映的群体杂合度有较大差异,其中位点PTR45在LZ群体的观测杂合度最高为0.9.6 7,位点PTR103b在HZ群体的观测杂合度最低为0.4.9 1。在某一个群体中的各位点的观测杂合度也差异较大,如在HZ群体,位点PTR33a的结果是Ho5.0.791 7,而位点PTR103b得到的仅为Ho5.0.409 1,表明用单个位点进行群体杂合度的检测,其结果并不一定能够代表群体的真实杂合度水平,通常取多个位点杂合度的平均值。根据所得的杂合度平均值可知,5个群体的平均期望杂合度相差不大,以LZ群体的最高He5.0.770 4,ZS群体的杂合度最低He5.0.728 3;平均观测杂合度在0.648.1 ~ 0.7.0 4之间。观察5个群体的观测杂合度和期望杂合度发现,两者均相差较小,说明每个群体内的等位基因频率都未偏离Hardy-Weinberg平衡。通过对5个群体中各位点的卡方检验可知,除了LZ群体和HC群体的PTR145位点,HZ群体的PTR98a和PTR112位点以及ZS群体的PTR98a和PTR103b位点表现为显著和极显著偏离Hardy-Weinberg平衡外,其余位点均符合Hardy-Weinberg平衡。
表5 三疣梭子蟹5个群体间的遗传相似性指数及遗传距离
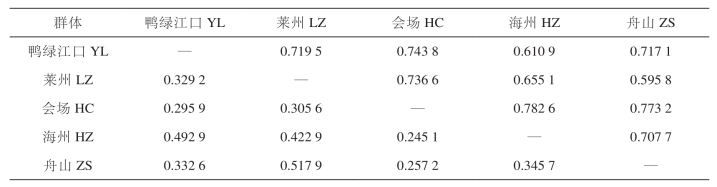
注:数字距阵对角线以上的数表示群体间遗传相似性指数,数字距阵对角线以下的数表示群体间遗传距离
2.1.3.5 遗传距离和聚类分析
通过遗传距离可以估计群体遗传分化程度,根据Nei(1972)的标准利用软件POPGENE计算种群间的遗传距离和遗传相似性指数,由表6可以看出,三疣梭子蟹5个群体间的遗传距离在0.245.1 ~ 0.5.7 9之间,遗传相似性指数从0.595.8 ~ 0.7.2 6,其中HC和HZ的遗传距离最小,相似性最高,LZ和ZS的遗传距离最远,相似性最小。根据遗传距离得出5个群体的UPGMA聚类树(图4),HC和HZ两个群体遗传距离最近先聚到一起,再与ZS群体相聚,之后与YL群体相聚,LZ群体与这4个群体的遗传距离较远,最后聚在一起。

图5 三疣梭子蟹5个群体的聚类分析图
2.1.3.6 群体遗传变异分析
Fst值可以用来测量群体间的遗传分化程度,采用软件FASTA对5个群体的遗传分化指数Fst进行计算(表6),8个位点的遗传分化指数在0.054.8 ~ 0.1.8 3之间。从Fst值的大小可以看出,5个群体间产生了中等程度的遗传分化(0.05 , Fst , 0.15),并且分化程度都达到了极显著水平。
表6 三疣梭子蟹5个群体之间的遗传分化指数(Fst)

注:*P , 0.05显著;** P , 0.01极显著
2.1.3.7 群体间的遗传结构变化
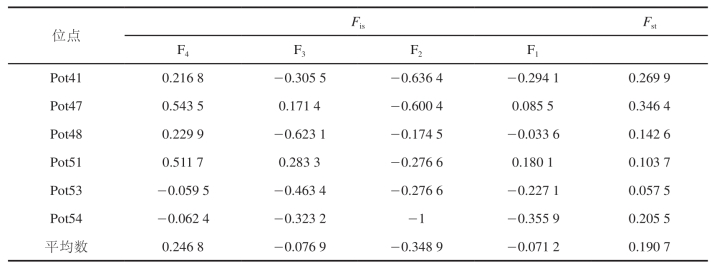
通过POPGENE软件对各家系进行F-检验,结果(表7)显示,根据遗传分化指数的界定,各家系之间存在轻度遗传分化(Fst , 0.05)的位点为0个;有中度遗传分化(0.05 , Fst , 0.15)的位点为8个;有较大遗传分化(0.15 , Fst , 0.25)的位点为3个;有很大遗传分化(Fst , 0.25)的位点为6个。Fst值均大于0.05,表明4个家系之间遗传分化较强。各家系的平均基因分化系数为0.1.0 7,表明19.07%的遗传分化来自群体间,80.93%的遗传分化来自群体内。另外,对Fis值的计算显示,4个家系在整体上均表现为一定程度的杂合子缺失,其中F4有4个位点、F3有10个位点、F2有13个位点、F1 有8个位点处于杂合子缺失状态。Fis值(近交系数,值大于零表示观测杂合子缺失,值小于零表明杂合子过剩,值等于1 时表明杂合体完全缺失)的计算结果表明,就平均数而言4个家系除F4代表现杂合子缺失外,其他家系均表现为一定程度的杂合子过剩。说明到了F4后,近交作用开始体现,使很多基因出现流失。
表7 16个微卫星位点在三疣梭子蟹4个家系中的F-分析
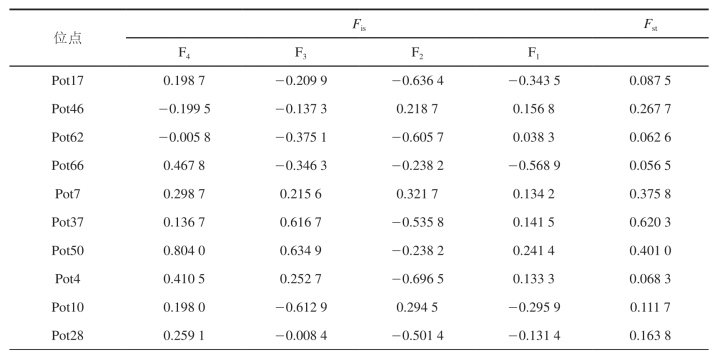
续表
采用Nei的方法计算群体间的遗传相似性和遗传距离,结果(表8)表明,最大遗传距离在F4与F3之间,为0.4.4 9,最小遗传距离在F3与F1之间,为0.1.7 8。F3与F1之间的遗传相似性为0.8.8 9,表明两个家系亲缘关系最近。
表8 三疣梭子蟹4个群体的相似性及遗传距离
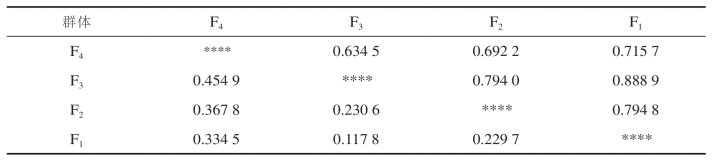
注:对角线以下为遗传距离。对角线以上为遗传相似性。
免责声明:以上内容源自网络,版权归原作者所有,如有侵犯您的原创版权请告知,我们将尽快删除相关内容。
















