第三节 其他表观遗传调控的研究方法
表观遗传学的研究内容包括了除基因序列突变外的DNA可遗传变化,或者与其相关的蛋白质。在表观遗传学这个领域中的许多研究者把目光聚焦在组蛋白修饰和DNA甲基化上,这两种相互关联和相互作用的分子机制都和表观遗传现象有关。很多方法分别用来研究表观遗传过程,而在过去的数十年中许多新进展层出不穷,它们被用来阐明表观遗传的遗传特征。遗传学与表观遗传学是遗传学研究的两个主要领域,在分析技术方面有许多交叉,可以互相借用。现将它们概述如下。
一、染色质免疫沉淀分析
染色质免疫沉淀(chromatin immunoprecipitation,ChIP)技术是一种在体内研究DNA和蛋白质相互作用的方法。在生理状态下把细胞内的DNA与蛋白质交联在一起,超声波将染色质打碎后,用所要研究的目的蛋白特异性的抗体沉淀这种交联复合体。只有与目的蛋白结合的DNA片段才能够被沉淀下来。应用PCR或DNA芯片等分子克隆技术,分析沉淀中的DNA,可以找出在生理条件下DNA结合蛋白与DNA序列结合的位点,从而反映体内基因表达调控的真实情况。
1.基本步骤 见图10-10。
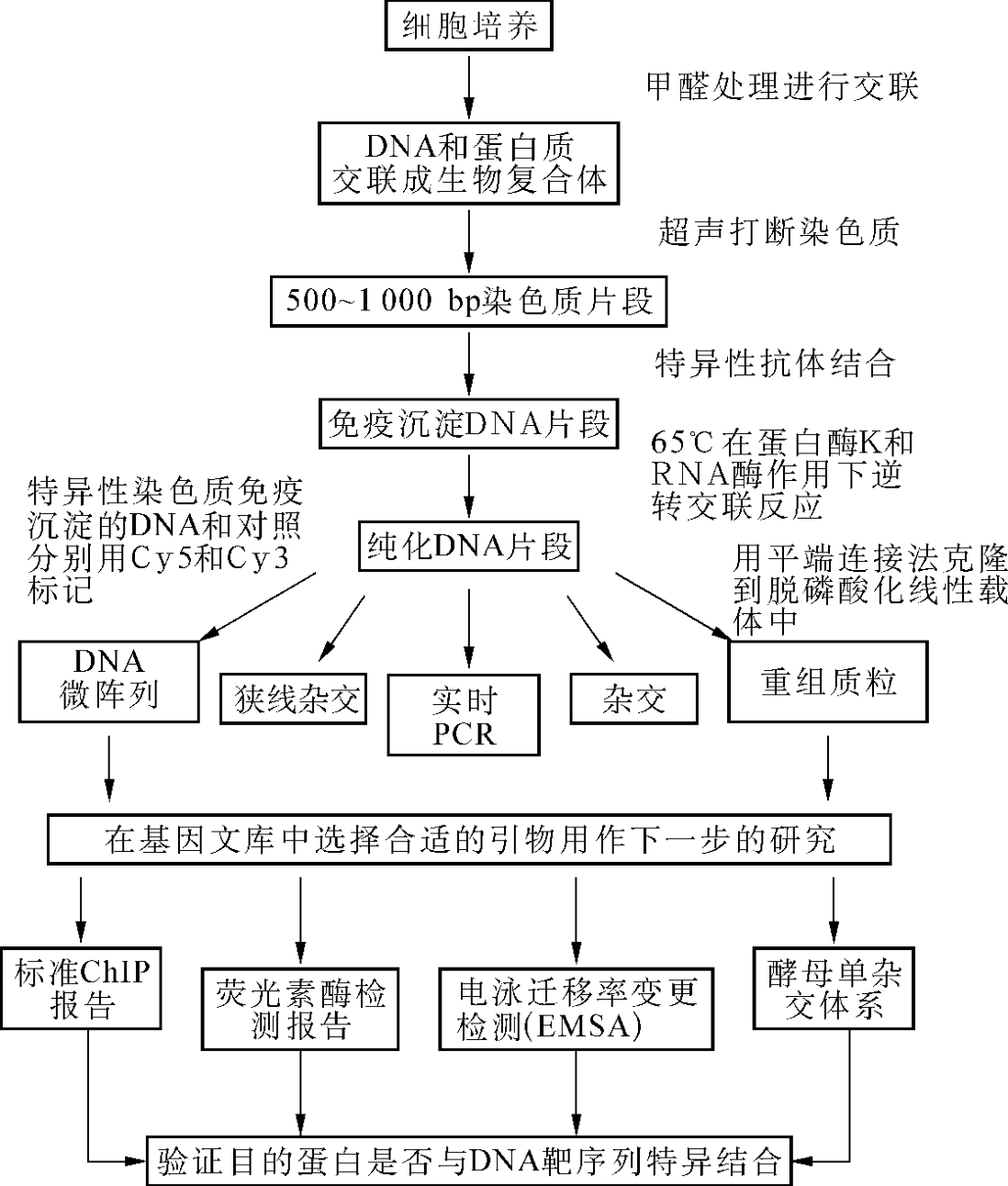
图10-10 染色质免疫沉淀技术示意图
(1)细胞的甲醛固定:甲醛是一种高分辨率的可逆的交联剂。能有效地使蛋白质-蛋白质,蛋白质-DNA发生交联。它能使DNA碱基上的氨基或亚氨基和蛋白质上的α-氨基及赖氨酸、精氨酸、组氨酸、色氨酸的侧链氨基与另外的DNA和蛋白质上的氨基或亚氨基交联在一起。在几分钟内形成生物复合体防止细胞内组分的重新分布。甲醛对游离的DNA双链不起作用,从而避免对DNA造成损伤。关键是甲醛的交联过程是完全可逆的,便于后续步骤中分别对DNA和蛋白质进行分析。通常的交联时间为5~60分钟,可以随时加入甘氨酸终止反应。
(2)超声处理:甲醛处理后的染色质对限制酶和DNaseⅠ有较强的抵抗力,因此通常使用机械剪切力使染色质断裂。打断后的染色质片段的平均长度约500bp(可通过琼脂糖凝胶电泳鉴定)。这样做的好处在于:①有利于染色质从裂解的细胞中溶解至提取液中;②对于蛋白质在DNA上结合位点的高分辨率作图非常重要。
(3)免疫沉淀:抗体必须与它的目的蛋白质在缓冲液及淋洗过程中呈特异性紧密结合。抗体的量要优化,防止非特异结合,可以通过预实验确定抗体的用量。用结合在涂脂糖凝胶或葡聚糖凝胶上的蛋白A或蛋白G回收免疫沉淀复合体。经过洗涤除去非特异结合的染色质后,用1%SDS+Na2CO3洗脱免疫沉淀复合体。
(4)交联反应的逆转和DNA的纯化:在免疫沉淀复合体中加入不含DNase的RNase和ProteinaseK(也可以不加蛋白酶K,交联逆转后用QIAquick DNA cleanup system回收DNA,蛋白还可用于进一步分析),65℃保温6小时,可使交联逆转。经酚氯仿抽提、乙醇沉淀纯化DNA。
(5)定量PCR:由于纯化得到的DNA一般只有几个纳克(ng),通常需要用PCR的方法扩增富集的沉淀DNA。引物的选择取决于实验目的,如若要鉴定目的蛋白质特异结合的位点,除必须设计一对能使扩增片段跨过该位点的引物外,至少还应设计一对扩增的DNA序列中没有目的蛋白质结合位点的对照引物。
PCR产物经凝胶电泳后可以用溴化乙啶或SYBR Green染色,也可以在扩增体系中加入少量放射性放射性核素,进行放射自显影。用实时PCR进行定量分析时可以直接观察到线性范围,及直接评判反应的质量。
2.基因芯片与染色质免疫沉淀技术 由于使用染色质免疫沉淀技术得到的结果是大量而又复杂的,因此运用普通方法对其结果的分析和处理就显得力不从心。近年来发展起来的基因芯片与染色质免疫沉淀技术相结合(ChIP on chip)的方法,大大促进了在基因组水平上高通量对DNA和蛋白质的相互作用的研究。
因为染色质免疫沉淀富集的DNA极少,所以需要一个PCR步骤来扩增。用LM-PCR(ligation-mediated PCR)的方法,可以使染色质免疫沉淀后所要研究的DNA(特异和非特异的染色质免疫沉淀产物)的比值在扩增以后不发生偏差。特异性染色质免疫沉淀的DNA和对照DNA的PCR产物分别用Cy5和Cy3荧光标记,用于与含有整个基因组的DNA芯片进行杂交。通过比较两种荧光信号的强度筛选出目的蛋白可能的结合序列,在基因组的水平上绘制出目的蛋白的分布图谱。
3.应用及发展前景 随着“组蛋白密码”的提出,各种修饰组蛋白的特异性抗体被开发出来,大大促进了组蛋白修饰与基因表达的研究。例如Li等人应用染色质免疫沉淀技术,发现组蛋白H3Lys4的乙酰化和甲基化可以激活Gnas基因的表达。而组蛋白H3 Lys9的甲基化和DNA的甲基化可以使Gnas基因表达沉默。Shan等人发现乙酰化的组蛋白与PCNA启动子的结合依赖于p53和电离辐射。Hashimshony等人发现DNA甲基化导致H4的脱乙酰化和H3Lys9的甲基化,阻止H3Lys4的甲基化。这在小鼠发育调控的染色质重构过程中发挥了重要的作用。
近年来ChIP经过不断的发展和完善,特别是与DNA芯片和分子克隆技术相结合,可用于高通量的筛选已知蛋白因子的未知DNA靶点和研究反式作用因子在整个基因组上的分布情况。此技术甚至也可以用于研究RNA与蛋白质的相互作用。具体方法与DNA的染色质免疫沉淀技术类似。在逆转交联以后,要纯化RNA,并经过反转录以便进一步分析或者纯化RNA后与cDNA芯片杂交。这种研究方法能比酵母三杂交系统更好的研究RNA与蛋白质的作用,因为它更接近于体内的真实情况。
二、全基因组定位技术
全基因组定位技术是一种将ChIP与基因表达的系列分析(serial analysis of gene expression,SAGE)相结合的方法。
SAGE是一种快速分析基因表达信息的技术。它通过快速和详细地分析成千上万个表达的序列标记片段(expressed sequence tag,EST)来寻找出表达丰度不同的SAGE标签序列,从而获得几乎全部基因组表达信息。将ChIP和长标签SAGE(long SAGE,SAGE的一种改进方法)相结合建立的GMAT,可用于测定全基因组中组蛋白修饰的分布状态(图10-11)。
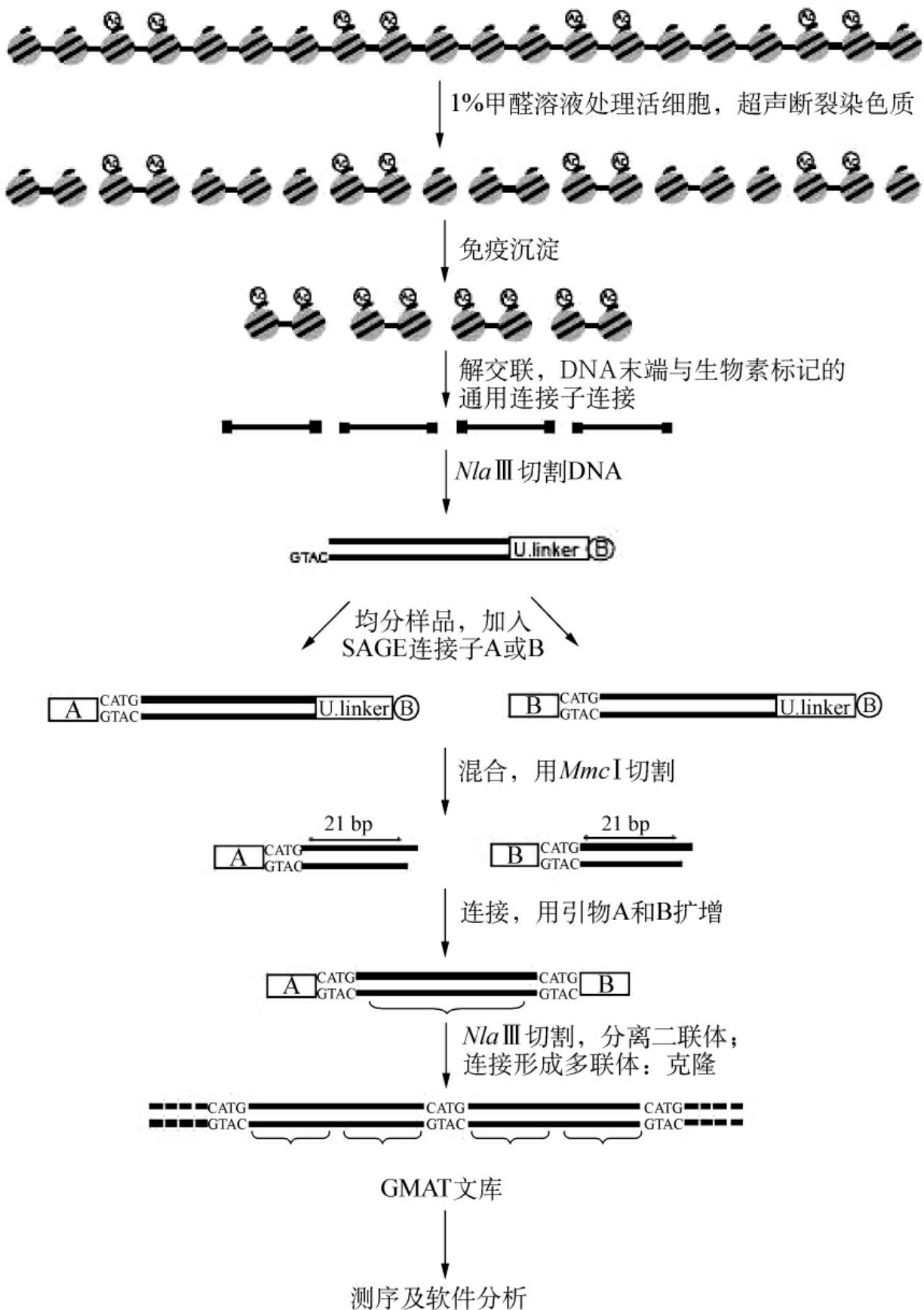
图10-11 GMAT原理示意图
1.主要步骤
(1)交联,用1%甲醛溶液处理活细胞,使DNA与组蛋白稳定结合。
(2)超声断裂染色质,使染色质断裂成300~500bp的片段。
(3)免疫沉淀,用特异抗体富集结合了DNA片段的修饰的组蛋白。
(4)酶交联。
(5)富集的DNA片段用Klenow酶补平,再与生物素标记的通用连接子连接。
(6)用链霉抗生物素蛋白珠子分离标记的DNA。
(7)用NlaⅢ消化,使DNA产生3′-GTAC(粘端)。
(8)用SAGE连接子A和B相连接,将样品DNA平分为两组,分别与均含有第二类限制性内切酶MmeⅠ识别位点,并具有3′-GTAC粘末段的SAGE连接子A和B连接。
(9)用MmeⅠ消化两组样品,获得一系列含21~22bp样品DNA序列标签的片段。
(10)将两组样品混合、连接。标签片段连接形成两端分别为A和B的二联体。
(11)以连接子A和B上的序列为引物,PCR扩增。
(12)用NlaⅢ切割,分离二联体标签,再连接形成500~1500bp的多联体标签。
(13)将多联体标签克隆到测序载体中,构建成GMAT文库。
(14)测序及软件分析,使用SAGE软件将21~22bp的标签序列从测序数据中提取出来,与基因组序列比对。GMAT文库中某标签出现的频率代表了结合在基因组对应位置上组蛋白抗原表位被抗体识别的多少,即目的修饰组蛋白在该段DNA序列上出现的频率。
2.应用 GMAT具有分辨率高、结果可信度高的优点,尤其适用于全基因组组蛋白修饰的定量测定。与ChIP on chip相比,GMAT不依赖于预先选择的序列,并且可以扫描全基因组。对各种修饰组蛋白进行GMAT分析,将有助于人们进一步认识“组蛋白密码”。这种方法同样可以应用于鉴定其他染色质修饰酶以及转录因子的作用位点。
三、体内DNA足迹法
体内DNA足迹法(in vivo footprinting),是检测体内蛋白质-DNA相互作用的重要方法。先用切割试剂(DMS、DNaseⅠ)处理细胞和细胞核,使基因组DNA产生随机切口。如果DNA特定区域上结合了蛋白质,那么这部分DNA就会受到保护而不被切割。断裂后的DNA经纯化,利用连接介导PCR(ligation-mediated PCR,LM-PCR),扩增出一系列长度不同并被标记的DNA片段,凝胶电泳后形成一系列梯带。比较先用切割试剂处理后再提取基因组DNA样品和先提取基因组DNA再用切割试剂处理样品(对照)结果间的差异。若实验组中缺少某些条带,则这些条带在基因组DNA上对应的位置即为蛋白质结合所形成的保护区。与体外DNA足迹法相比,体内DNA足迹法能真实反应体内蛋白质在DNA上的结合位点(图10-12)。
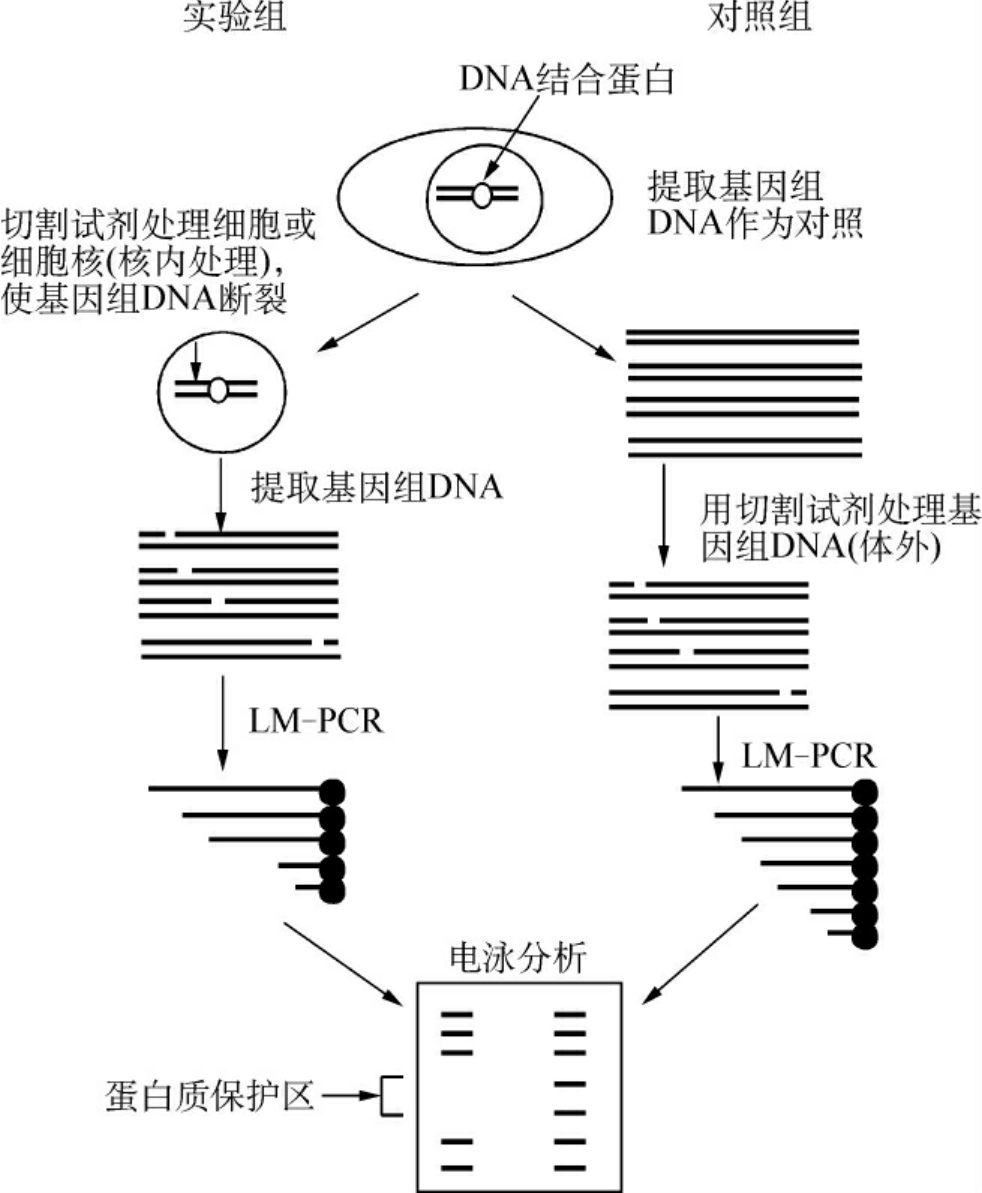
图10-12 体内DNA足迹法原理示意图
1.主要步骤
(1)用切割试剂处理细胞或细胞核:常用的切割试剂有硫酸二甲酯(DMS)和DNA酶Ⅰ(DNaseⅠ)。DMS可使基因组DNA中的G和A发生甲基化修饰,随后用哌啶处理使DNA在修饰处断裂。用DNaseⅠ处理细胞可使基因组DNA随机断裂。
(2)提取并纯化基因组DNA:经过抽提、RNA酶A消化、乙醇沉淀等纯化基因组DNA。
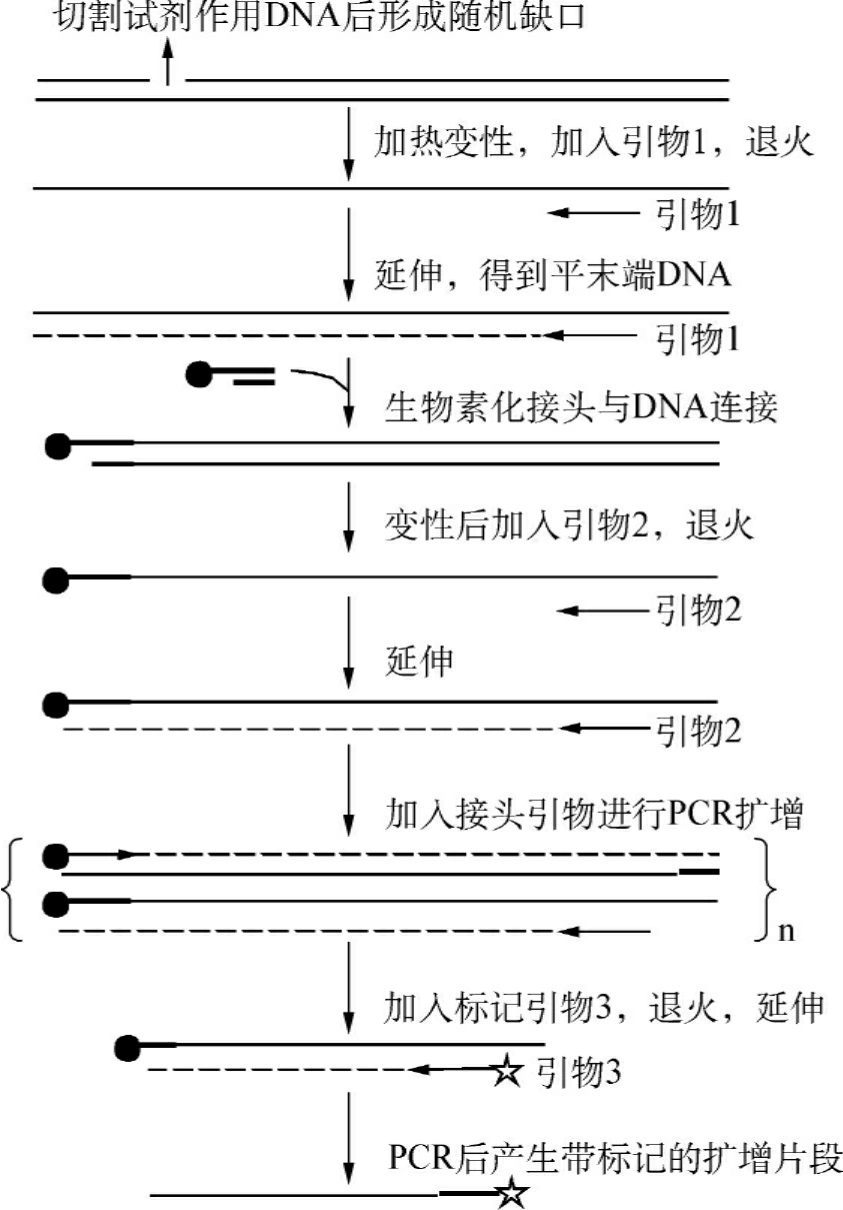
图10-13 LM-PCR原理示意图
(3)进行LM-PCR:见图10-13。
1)DNA变性后加入引物1,退火。引物1一般距离目标区域至少100bp;DNA聚合酶催化下延伸得到平末端的双链DNA。
2)加入单向接头(生物素化),DNA连接酶催化平末端DNA片段与接头相连。接头是由两段互补的长短不同的寡聚核苷酸退火形成的部分双链DNA,一端为平端,一端为粘端。为防止接头自连,两个片段的5′端均缺少磷酸,连接时只有较长片段的3′羟基可与基因组DNA平端5′磷酸连接。
3)加入特异引物2,延伸后仍获得平末端双链DNA,引物2比引物1更靠近内侧(引物1的3′端),以保证扩增的特异性。接着加入接头引物,其序列与接头中较长的片段互补。PCR扩增一定的循环。在反应体系中加入标记的引物3后,进行第二轮PCR。引物3可用放射性核素32 P标记,位置比引物2更靠近内侧。
4)纯化DNA,聚丙烯酰胺凝胶电泳,放射自显影或磷屏分析。
2.应用 体内DNA足迹法可直接用于对基因调控区的初步研究,与其他方法相比具有分辨率高、更能反应体内的真实情况的特点。还可用于验证体外实验的结果。如某调控元件在体外使用纯化的重组蛋白出现了条带,而用体内法也出现相同的条带,那么可以证明这种蛋白确实在体内发挥了作用。另外,它还可以用来研究内源性可诱导的或具有细胞特异性的表达调控。因此,体内DNA足迹法是研究基因转录调控的有力工具。
四、电泳迁移率变更检测法
凝胶迁移或电泳迁移率实验(electrophoretic mobility shift assay,EMSA)是一种研究DNA结合蛋白和其相关的DNA结合序列相互作用的技术,可用于定性和定量分析。EMSA最初用于研究DNA结合蛋白,目前已用于研究RNA结合蛋白和特定的RNA序列的相互作用。通常将纯化的蛋白和细胞粗提液和32 P放射性核素标记的DNA或RNA探针一同保温,在非变性的PAGE上,分离复合物和非结合的探针。DNA-复合物或RNA-复合物比未结合的探针移动得慢(图10-14)。该图,标记了的DNA由于与同一种细胞蛋白结合,于是在凝胶电泳中移动速度变慢,放射自显影后呈滞后的条带。放射性核素标记的探针依研究的结合蛋白的不同,可以是双链或者是单链。竞争实验中采用含蛋白结合序列的DNA或RNA片段和寡核苷酸片段(特异)和其他非相关的片段(非特异),来确定DNA或RNA结合蛋白的特异性。
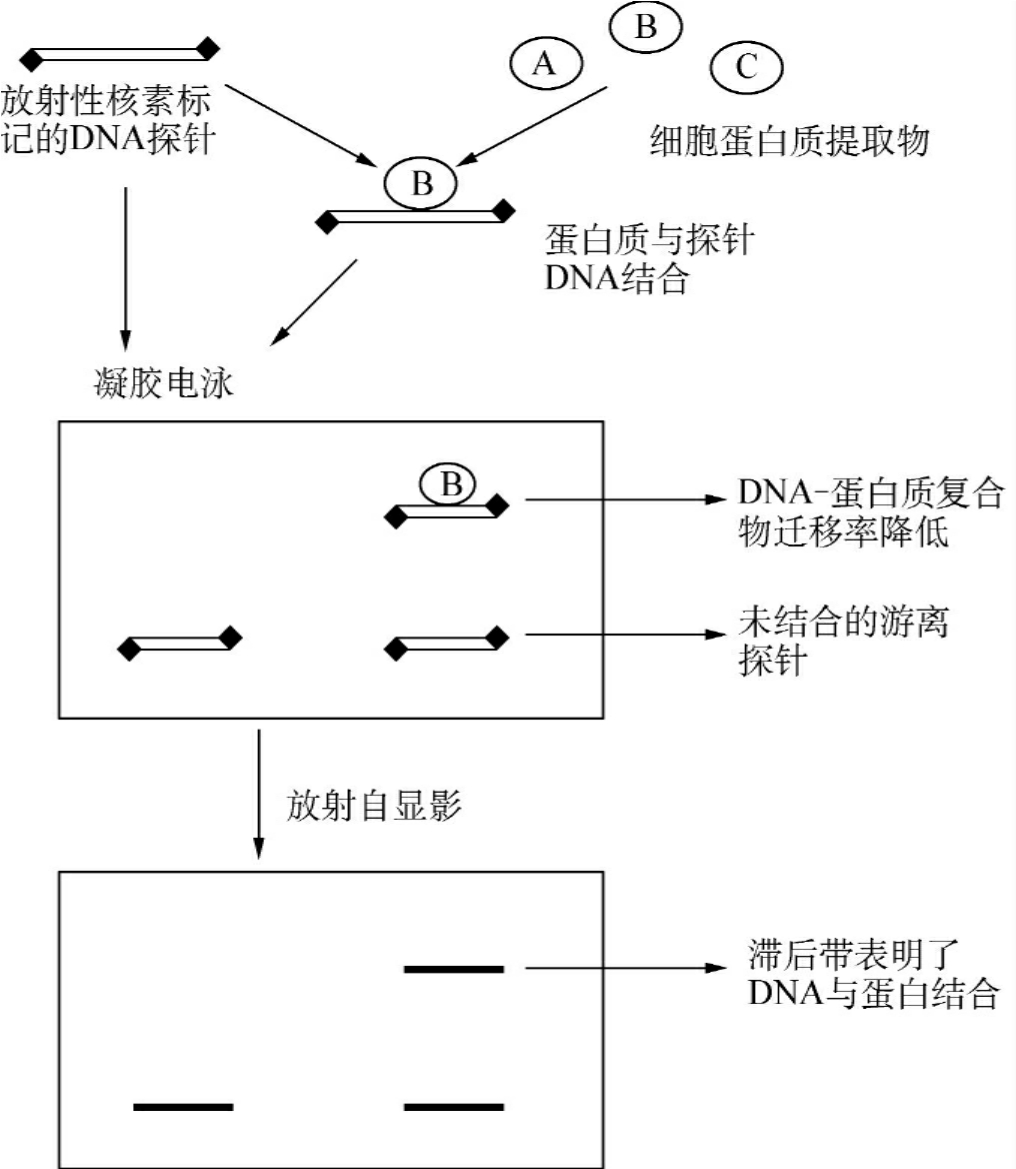
图10-14 EMSA基本原理
采用EMSA可以鉴定目的基因的调控序列中是否存在特定的DNA结合蛋白位点,也可以鉴定特定的细胞核蛋白中是否存在与已知的顺式调控元件结合的蛋白质。
1.基本步骤
(1)制备含有特异结合蛋白位点的双链DNA探针。探针的长度一般控制在25~100bp为宜,若片段过长则在PAGE中不易与DNA-蛋白质复合物分开,而且容易造成含有多种DNA结合蛋白位点,干扰对结果的分析。
(2)核蛋白与DNA探针的结合反应
1)由于核蛋白提取物中存在许多非特异DNA结合蛋白,为此在反应体系中加入适量的鲑精DNA或poly(dI:dC),作为非特异竞争剂。
2)为验证DNA与蛋白质结合的特异性,在反应体系中加入过量的非标记的DNA探针,与标记探针竞争结合蛋白质。若导致结果放射自显影滞后带消失,那么证明目的蛋白与标记探针是特异结合的。
3)确定复合物中蛋白的特征可能会困难,但有一些方法做这方面的研究。如有目的蛋白的抗体,可进行超迁移实验,抗体和蛋白-探针复合物中的蛋白结合,使复合物的迁移延迟,形成超迁移。增量的抗体加入到结合反应中。抗体可在蛋白和探针反应后加入,也可将抽提物与抗体结合后,再加入探针。取决于抗体的特定的抗原决定簇,前者有利于超迁移复合物的形成,后者阻止复合物的形成导致原复合物的强度的减少。
(3)PAGE:聚丙烯酰胺的浓度一般为6%(30∶1丙烯酰胺:双叉),在特定条件下可用高或低的浓度。大多数蛋白用10~15V的电压电泳,解离快的蛋白用短时间和高的电压(30~35V),电泳时所用的TBE和TAE必须是新配制的,无沉淀。加样缓冲液中的色素会导致不稳定复合物的解离,应用不含考马斯蓝和二甲苯蓝的加样缓冲液。当带型不紧密出现拖尾时,表明复合物存在解离。如复合物不进入凝胶则表明所用的蛋白或探针过量,或盐的浓度过量不适用于这一反应。目前可用高强度琼脂糖凝胶分离蛋白-探针复合物。
(4)放射自显影观察结果:见图10-15。
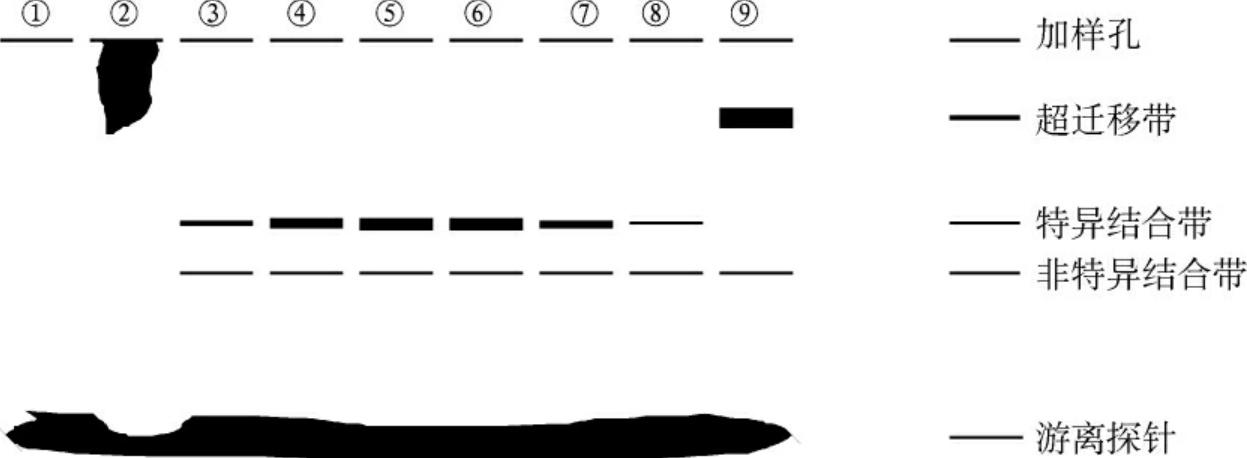
图10-15 电泳迁移率改变示意图
注:①探针;②探针与核蛋白结合;③~⑤非特异性竞争,固定探针和核蛋白量,逐渐增加鲑精DNA;⑥~⑧特异性竞争,固定探针、核蛋白和鲑精DNA量,逐渐增加未标记特异DNA量;⑨抗结
合蛋白的抗体作用后,形成抗体-蛋白质-DNA三元复合物,其迁移率进一步降低,即“超迁移”
2.发展前景 检测转录因子的方法有免疫组化、Western blot、EMSA等。免疫组化、Western blot虽然操作相对简单,无放射性损伤,但特异性不强,不能反映核蛋白在核内的DNA结合活性。而某些核蛋白的抗体价格相当昂贵,限制了应用。因此,目前检测核转录因子活力最常用的方法仍为EMSA。
现已知道,结构基因组学已经很容易实现高通量分析,但在功能研究这个层次上,目前还没有真正高通量的分析技术出现。技术的缺乏是后基因组学时代功能研究的主要瓶颈。和其他功能研究技术一样,EMSA本身也存在诸多缺点,比如对于低亲和力结合很难进行鉴定;难以比较不同片段之间亲和力大小的差异;无法得知结合位点的具体位置。很难真正重建体内蛋白质与DNA之间结合过程。对EMSA进行改良或发展更好的研究蛋白质与DNA互相作用的技术很有必要。
五、染色质构象捕获技术
染色质构象捕获技术(chromosome conformation capture,3C)是用于确定任何两个基因组之间相互接触频率的方法之一。通过测定在同一个或不同染色质上的位点之间相互作用的频率,从而揭示它们的相对空间排列,并提供有关染色质纤维的物理性质和构象的信息。常用甲醛固定细胞,使全基因组相邻蛋白质与蛋白质及蛋白质与DNA之间的物理性接触的节段发生交联;然后用限制性内切酶酶切及用连接酶将断开的片段进行连接,使在临近的片段之间产生新的连接位点;再对新产生的连接产物进行PCR定量分析。需要指出的是并非所有能产生新的PCR产物的酶切片段在核内都能发生具有功能性意义的相互作用。有时相距较远的片段被交联起来只是由于它们在核内相互接近,可能在交联过程中发生相碰所致。
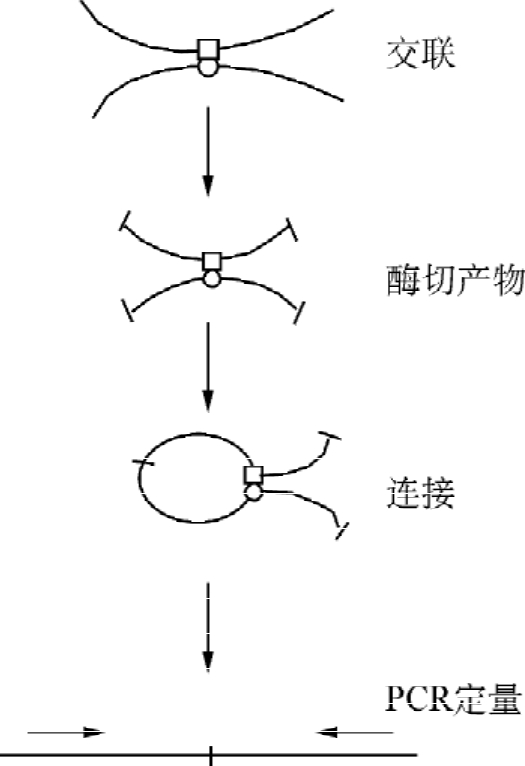
图10-16 染色质构象捕获技术流程图
染色质构象捕获基本步骤如图10-16所示。
(1)固定:用甲醛固定细胞,使全基因组中相邻蛋白质与蛋白质及蛋白质与DNA之间的物理性接触的节段发生交联。甲醛固定的条件是3C技术的关键。
(2)裂解细胞,限制性内切酶消化:根据拟分析的染色质所在区域选择合适的内切酶。
(3)连接:必须充分稀释DNA,以利于分子内连接,即只有已交联的内切酶片段发生连接反应。
(4)解交联:进行解交联及纯化DNA。
(5)PCR扩增:为提高灵敏度可采用巢式PCR方法,设计的引物应预先在含有等分子数的、可能的连接产物的DNA样本上进行检测。
(谢佐俊 潘銮凤)
参考文献
1.薛京伦.表观遗传学-原理、技术与实践.上海:上海科学技术出版社,2006
2.沈羽琲.染色质与表观遗传调控.北京:高等教育出版社,2006
3.董玉玮,侯进慧,朱必才等.表观遗传学的相关概念和研究进展.生物学杂志,2005,22:1~3
4.朱静.染色质的重塑与基因表达调控.国外医学·临床生物化学与检验学分册,2005,5:12~14
5.李珺,薛丽香,张宗玉等.组蛋白密码.医学分子生物学杂志,2004,1:100~103
6.沈金花.组蛋白密码与表观标志.国外医学·分子生物学分册,2002,24:337~340
7.宋春雷,刘红林.组蛋白的泛素化和去泛素化修饰.细胞生物学杂志,2007,29:51~55
8.黄世杰.癌的表观遗传性治疗:过去,现在和将来.国外医学·药学分册,2006,5:342~344
9.基因组医学、染色体组和人类疾病基因(5).现代临床医学生物工程学杂志,2004,10:442~449
10.庄海滨,朱景德,刘湘军.人类全基因组范围的CpG岛的预测与分析.生物物理学报,2006,22:351~359
11.顾婷婷,张忠明,郑鹏生.DNA甲基化研究方法的回顾与评价.中国妇幼健康研究,2006,6:555~560
12.王春雨,石建党,朱彦,张琚.染色质免疫沉淀技术在研究DNA与蛋白质相互作用中的应用.遗传,2005,27:801~807
13.蔡磊.差异表达基因的几种筛选方法.第四军医大学学报,2007,28:286~288
14.Minoru Toyota,Coty Ho,Nita Ahuja,et al.Mutsumi Ohe-Toyota,Stephen B.Baylin,and Jean-Pierre J.Issa.Identification of differentially methylated sequences in colorectal cancer by methylated CpG island amplification1.Cancer Res,1999,59:2307~2312
15.Tina Bianco,Damian Hussey,and Alexander Dobrovic.Methylation-sensitive,single-strand conformation analysis(MS-SSCA):a rapid method to screen for and analyze methylation.Human Mutat,1999,14:289~293
16.Edward J.Oakeley DNA methylation analysis:a review of current methodologies.Pharm Therap,1999,84:389~400
免责声明:以上内容源自网络,版权归原作者所有,如有侵犯您的原创版权请告知,我们将尽快删除相关内容。

















