DNA的复制过程十分复杂。目前对于复制的了解主要来源于原核生物。DNA复制是连续的过程,为描述方便,把它分为起始、延长和终止3个阶段。
(一)起始阶段
1.起始复合物(initiation complex)的形成 原核生物例如大肠埃希菌DNA的复制是从固定的复制起始点开始。我们把这一起始点称为ori C(origin C)。大肠埃希菌ori C的DNA片段约含245个碱基对,其中有两个区域在复制起始中起关键作用,一处是9个核苷酸的5次重复结构,另一处是13个核苷酸的3次重复序列(图3-7)。ori C中9-核苷酸重复序列可以被Dna A蛋白识别并结合形成含有20~30亚基的起始复合物。与9-核苷酸重复序列结合的Dna A可促ori C中的13-核苷酸重复序列局部发生解链,形成解链复合物。这一过程的确切机制尚不清楚。由于Dna A并不具有解链的酶活性,推测可能与Dna A结合后引入的扭转力有关。
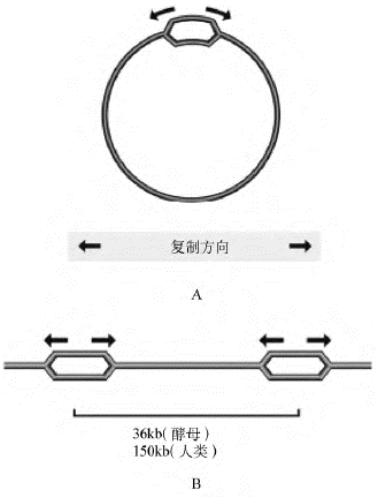
图3-6 环状细菌染色体(A)和线形真核染色体(B)复制
2.引发前体(prepriming complex)的形成 与9-核苷酸重复序列结合的Dna A引导了另外两种蛋白质Dna B/Dna C的复合物进入局部解链区,形成引发前体复合物。Dna C的作用非常短暂,很快就从引发前体复合物上脱落,它的作用可能是协助Dna B的结合。Dna B是一种解链酶,能使双链DNA进一步解链(图3-8)。
解链是一种高速的反向旋转,其下游势必发生打结现象,此时DNA拓扑异构酶起到调节作用,促进复制叉的不断解链。双链解开后,SSB结合到开放的单链上,起稳定和保护单链模板的作用,同时有利于参与DNA链延长的各种酶结合到打开的单链上,开始DNA复制过程的延长阶段(图3-9)。
3.引物(primer)的合成 复制过程需要引物,引物是由引发酶(primase,即Dna G蛋白)催化合成的短链RNA分子(图3-10)。引发酶是一种RNA聚合酶,它又不同于催化转录过程的聚合酶。高度解链的模板与Dna B/Dna C蛋白复合体促进引发酶加入进来,合成引发体(primosome)。引发体的蛋白质部分在DNA链上可以移动,并需要由ATP供给能量。引发体到达适当位置后,以4种NTP为原料,以解开的DNA链为模板,按5′→3′方向合成RNA引物。不同生物RNA引物的长短不同,从十数个至数十个核苷酸不等。引物末端游离的3′-OH成为进一步合成DNA的起点。在DNA聚合酶Ⅲ的催化下,由酶的β亚基辨认引物,第一个脱氧核苷酸加到引物的3′-OH上,形成磷酸二酯键,新DNA链的合成即已开始。
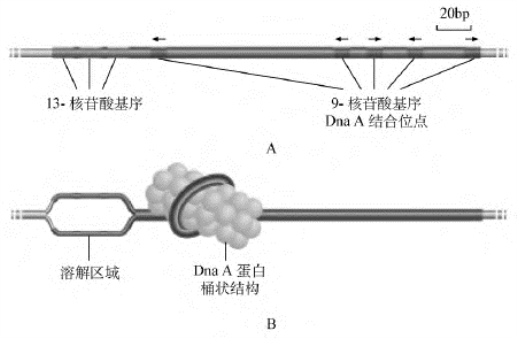
图3-7 大肠埃希菌复制起点
A.大肠埃希菌复制起点即ori C,长约245bp,包括3个13-核苷酸的重复基序,共同序列为5′-GATCTNTTNTTTT-3′,这里的N代表任何核苷酸,5个拷贝的9-核苷酸重复基序,共同序列5′-TTATTAC CAAC A-3′。这13-核苷酸的重复基序顺式重复地依次排列于ori C的一端。而9-核苷酸序列以顺式或反式重复的形式位于ori C的不同位置,这其中的3个重复序列,即从ori C左手末端数过来的第1、3、5个重复序列,被认为是Dna A结合的主要位置;另两个为次要位置。在所有的细菌中,复制起点在结构上相同并且重复序列也没有大的改变。B.Dna A蛋白与ori C结合模型,这种结合导致在AT富集的13-核苷酸序列处的双螺旋解开

图3-8 在大肠埃希菌DNA复制过程中Dna B解旋酶的作用
Dna B是一种5′→3′解旋酶,能够沿后滞链移动,同时使碱基对断裂。它与DNA拓扑异构酶共同作用解开双螺旋。为避免混淆,与Dna B结合的引发酶在图中未显示
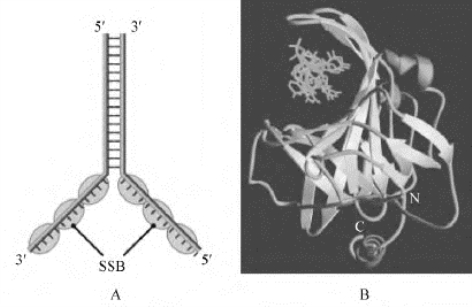
图3-9 DNA复制中单链结合蛋白(SSB)的作用
A.SSB可以与由解旋酶所产生的未配对的多聚核苷酸结合,从而阻止与其他链发生配对并能防止单链特异的核酸酶的降解;B.真核生物的SSB(称为RPA)的晶体结构。这种蛋白质所含有的β片层结构形成DNA(从末端观察)可以结合的孔道
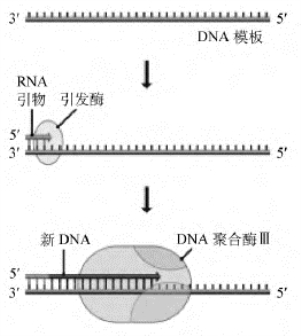
图3-10 细菌DNA 合成引发
示其合成RNA引物及其后的几个DNA核苷酸
(二)DNA链的延长
DNA链的延长是在DNA聚合酶(DNA polymerase,DNA pol)催化下,以四种三磷酸脱氧核苷(dNTP)为原料进行的聚合反应。聚合反应自引物的3′-OH端开始,以5′→3′方向逐个加入脱氧核苷酸dNTP,脱下焦磷酸PPi,使DNA链得以延长。原核生物DNA链的延长速度很快,在大肠埃希菌中以每秒500个脱氧核苷酸聚合速度进行。有几种不同的DNA聚合酶和连接酶参与DNA复制过程中链的延长,下面分别进行介绍。
1.DNA聚合酶Ⅰ DNA聚合酶(DNA polⅠ)的全称是依赖DNA的DNA聚合酶(DNA dependent DNA polymerase)。20世纪50年代末,Kornberg首次从大肠埃希菌中提取出DNA polⅠ,并在实验中证实加入模板、引物和底物,提取出来的DNA聚合酶可催化新链DNA的生成。这是一个具有重大意义的发现。当时曾把这种酶称为复制酶(replicase),以后随着其他种类DNA聚合酶的发现,这种最先发现的酶就被称为DNA polⅠ。DNA polⅠ有三种功能。
(1)5′→3′聚合作用,即在模板指导下,以dNTP为原料,在DNA或引物RNA的3′→OH末端逐个加上脱氧单核苷酸,形成3′,5′磷酸二酯键,使DNA链沿5′→3′方向延伸。
(2)3′-5′外切酶活性,即能从DNA链的3′端开始沿3′→5′方向进行水解反应,产生5′单核苷酸。在DNA复制时,当3′末端出现错配的碱基时,DNA polⅠ的3′→5′外切酶活性能识别和切除错配的碱基,纠正复制过程中的错误,保证DNA复制的正确性,因而具有校对功能。
(3)5′→3′外切酶活性,即能从5′→3′方向水解DNA链,产生5′单核苷酸。因此该酶参与RNA引物的切除。另外它还能跳过几个核苷酸,切除错配的核苷酸,起到修复作用,因此5′→3′外切酶活性可能在DNA的损伤修复中起作用。
此外,当DNA单链有缺口时,DNA polⅠ的5′→3′外切酶活性作用可以与聚合作用同时发生。切口处产生的3′末端可作为引物,在DNA polⅠ的5′→3′聚合活性的催化下,以互补的DNA单链为模板,依次将dNTP连接到切口的3′→OH末端,合成新的DNA单链;同时DNA polⅠ的5′→3′外切酶活性在切口处将旧链从5′逐步切除,所以缺口沿着合成方向在DNA分子上移动,这种移动称为缺口平移(nick translation),是分子生物学中一项重要的实验技术(图3-11)。
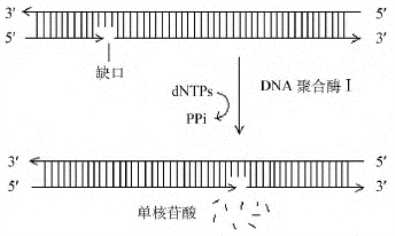
图3-11 缺口平移
用特异的蛋白酶可以把DNA polⅠ水解为大、小两个片段。小片段具有5′→3′外切酶活性,大片段(又称为Klenow片段)具有3′→5′外切酶活性和聚合活性。Klenow片段是进行分子生物学研究的常用工具酶。DNA polⅠ的聚合反应可以连续进行,但DNA链延长20个核苷酸后,酶就脱离了模板,故反应属于中等程度的连续聚合反应。DNA polⅠ催化的聚合反应速度慢,每秒钟约加入10个核苷酸。实际上,DNA polⅠ在大肠埃希菌中的主要功能不是催化DNA的复制,而是切除引物,填补冈崎片段间的空隙以及DNA损伤的修复。
1969年,DeLucia和Cairns分离到一株大肠埃希菌其DNA polⅠ的活性仅为正常大肠埃希菌的1%。这株突变菌的生长速度与正常菌无明显差异,但是在紫外线照射后,突变菌极易死亡。他们推测突变菌DNA复制功能正常,但DNA损伤后的修复机制缺失。这就意味着DNA polⅠ在DNA合成过程中的聚合作用可能是次要的,而它的外切酶活性在DNA损伤修复中发挥了主要作用。他们提出可能有不同于DNA polⅠ的聚合酶的存在,它负责DNA合成过程中的聚合反应。这一推测随后导致了DNA聚合酶Ⅱ和Ⅲ的发现。
2.DNA聚合酶Ⅱ DNA聚合酶Ⅱ(DNA polⅡ)具有5′→3′DNA聚合酶活性及3′→5′外切酶活性,只是在没有polⅠ和polⅢ的情况下起作用。它在体内的生物学功能还不完全清楚,可能与DNA的损伤、修复有关。
3.DNA聚合酶Ⅲ DNA聚合酶Ⅲ(DNA polⅢ)是由α、β、γ、δ′、ε、θ、τ、χ及φ等10种亚基组成的不对称二聚体。全酶的相对分子质量是400 000。其中,α、ε、θ组成核心酶。α亚基具有催化合成DNA的功能,ε亚基有3′→5′外切酶活性。两边的β亚基起着夹住模板又能使酶沿模板链滑动的作用。每一亚基的具体功能尚在研究中。
DNA polⅢ催化的聚合反应具有高度连续性,可以沿模板连续地移动,一般在加入5 000个以上的核苷酸之后才脱离模板,因此其催化的聚合反应速度快,大约每秒加入1000个脱氧核苷酸,在原核生物细胞内是真正起复制作用的酶。其3′→5′外切酶活性可阻止错误核苷酸的进入或除去错误的核苷酸,然后连续加入正确的核苷酸,因而具有编辑和校对功能。polⅢ与polⅠ协同作用可使复制的错误率大大降低,从10-4降为10-6或更低。
DNA polⅢ可以分别催化前导链和随从链的合成,自引物的3′-OH端开始,沿5′→3′方向逐个地加入脱氧核苷酸,使DNA链得以延长(图3-12)。复制叉前进时,前导链几乎可以不间断地延长,而随从链是分段(冈崎片段)合成的。合成冈崎片段时,当DNA链延长至下一个引物前方,DNA polⅠ立即发挥5′→3′外切酶功能,切除引物,并继续延长DNA链,填满切除引物后形成的空隙。最后,DNA连接酶通过生成磷酸二酯键将两个片段连接起来,封闭缺口,成为完整的随从链。
4.DNA连接酶(DNA ligase) 能催化双股DNA中一股缺口的3′-OH与5′-磷酸形成磷酸二酯键。连接反应中的能量来自ATP(或NAD+)。连接酶先与ATP作用,以共价键相连生成“酶-AMP”中间体。中间体与一个DNA片段的5′-磷酸末端相连接形成“酶-AMP-P-5′-DNA”。继而,又与另一个DNA片段的3′-OH末端作用,酶及AMP脱下,两个DNA片段以3′,5′磷酸二酯键相连接。通过连接酶的催化作用,随从链的各个DNA片段均可以3′,5′磷酸二酯键连接,生成一条DNA长链。
(三)DNA复制的终止

图3-12 DNA聚合酶Ⅲ催化DNA前导链和滞后链平行合成的模型
A.DNA聚合酶Ⅲ形成二聚体,滞后链通过DNA聚合酶Ⅲ形成环状结构,这样当聚合酶二聚体沿DNA分子进行复制时,前导链和滞后链可以同时复制。B.大肠埃希菌DNA复制过程中,相邻的冈崎片段相互连接时发生的事件:DNA聚合酶Ⅲ不具有5′→3′外切核酸酶活性,当它到达下一个冈崎片段末端时就停止DNA合成。这时,DNA合成由具有5′→3′外切核酸酶活性的DNA聚合酶Ⅰ继续,并与RNaseH共同作用去除RNA引物,代之以DNA。DNA聚合酶Ⅰ脱离模板前同样也取代部分冈崎片段DNA,遗留一个磷酸二酯键。这个磷酸二酯键由DNA连接酶连接,从而完成复制过程
复制的终止与DNA分子的形状有关。对于线性DNA来说,当复制叉到达分子末端时,复制即终止。一般说来,DNA链复制的终止不需要特定的信号。对于环状DNA来说,其复制形式多为双向复制,两个复制叉向不同方向行进,同时到达一个特定部位,也可能其中一个复制叉先到达此处停止,“等待”另一个移动较慢的复制叉,先行到达的复制叉并不会越过这一特定部位继续复制,这意味着此处有一个特异的终止信号。大肠埃希菌的两个复制叉的终止一般发生在位于ori C的相对处的区域,称为终止区(termination region,ter)。目前已发现至少6个ter序列,分别称为terE、terD、terA和terC、terB、terF,它们分别位于复制叉汇合点两侧约100kb处,terE、terD、terA是一个复制叉特异的终止位点,terC、terB、terF是另一个复制叉特异的终止位点,每个复制叉必须越过另一个复制叉的终止位点才能到达自己的终止位点。6个ter序列中都含有1个23bp的共有序列(GTGTGGTGT)。Tus蛋白(terminator utilization substance)识别和结合于终止位点的23bp共有序列,具有反解旋酶(contra-helicase)的活性,能阻止Dna B蛋白的解旋作用,从而抑制复制叉的前进,促使复制的终止。DNA复制完成后,又可在拓扑酶的作用下,将DNA分子引入超螺旋结构,进行进一步的装配(图3-13)。
DNA复制的过程非常复杂,由多种酶和蛋白质的参与,各自发挥不同的作用。现将大肠埃希菌DNA复制过程中参与的酶与蛋白总结于表3-1。
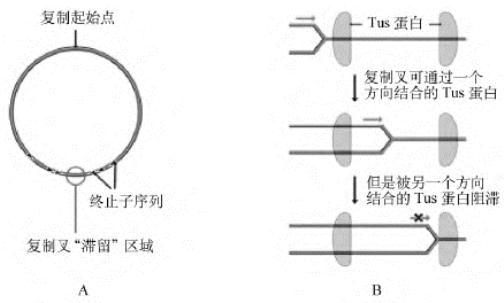
图3-13 大肠埃希菌中终止子序列的作用
A.大肠埃希菌基因组中6个终止子序列的位置,箭头方向显示复制叉所能通过的终止子序列的方向。B.Tus蛋白的结合,使复制叉能从一个方向通过,但从另一方向则不能。本图显示了左侧的Tus结合后,由于Dna B解螺旋酶能破坏Tus,从而使复制叉前进通过Tus;但将被第二个Tus所阻止,因为这时复制叉将面对Tus的β折叠链所形成的不可穿透的墙
表3-1 大肠埃希菌DNA复制相关的主要蛋白质

免责声明:以上内容源自网络,版权归原作者所有,如有侵犯您的原创版权请告知,我们将尽快删除相关内容。















