将特异条带回收,经相应SRAP引物扩增回收的模板DNA连接转化、培养,提取质粒,酶切确定目的片段后测序。抗高氨氮性状相关特异分子标记测序后,获得序列长度在166~571 bp间,与预期片段大小基本一致。抗高pH性状相关特异分子标记测序获得序列长度在104~1.0.3 bp间。BLAST比对发现获得的氨氮或pH特异片段序列与数据库中序列相似性均较低(一般小于15%),未找到同源性较高的功能基因组。
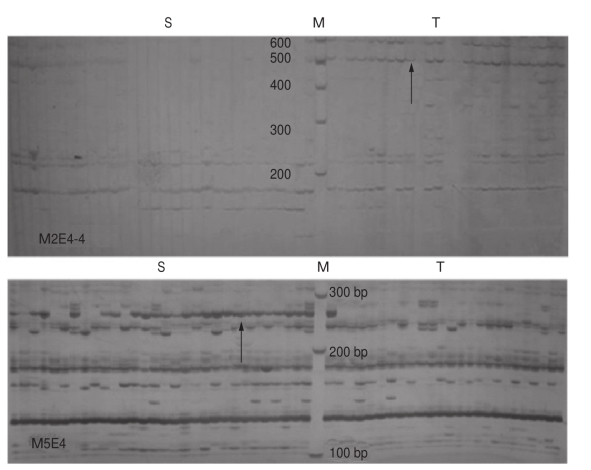
图23 SRAP引物扩增获得特异条带(箭头所示)
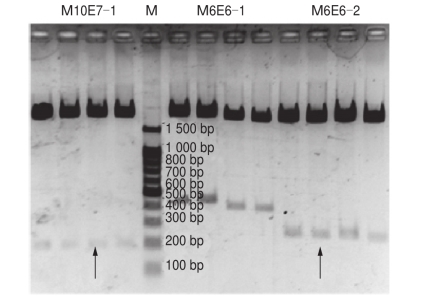
图24 部分特异片段质粒DNA酶切结果
表36 中国对虾抗逆性状相关特异标记的克隆与测序

续表
Michelmore(1991)提出了群体分析法(BSA)用于快速筛选与特定基因或者染色体区域连锁的分子标记。一般寻找与某一性状相连锁的分子标记的方法通常是分子标记技术结合BSA法或近等位基因系分析法。但由于中国对虾的一年生生物学特性,无法进行回交,并且目前构建的中国对虾连锁图谱的精密度和饱和度还不够高,因此利用BSA法是获得与目标性状连锁的分子标记的快捷途径(张天时等,2007)。SRAP标记技术自提出后已在植物研究领域获得广泛应用,融合传统分子标记技术(如RAPD、AFLP等)优点表现出简便、稳定、产率高、信息量丰富等技术特点,但目前该技术在水产动物相关研究中应用甚少。本研究将SRAP技术与BSA法相结合,依据标记在敏感组和耐受组中出现的频率及变化规律,共筛选出6个与抗高氨氮性状相关的特异性遗传标记,占总位点数的0.52%,标记大小在166~571 bp之间;7个与抗高pH性状相关标记,占总位点数的0.45%,标记大小自104 bp至1.0.3 bp。不足1%的特异标记频率发生改变也说明,本研究进行抗高氨氮和抗高pH群体选育的潜力还很大,可为进一步选育研究提供丰富的材料。
研究表明(Le et al,2000),氨氮胁迫影响对虾免疫相关基因的表达。细角滨对虾暴露于氨氮溶液后,酚氧化酶原和细胞黏附蛋白的转录编码量下降了50%~60%。SRAP的鲜明特点之一是依靠特定设计的引物结合基因组开放阅读框进行PCR扩增。本研究利用SRAP对获得特异条带序列进行分析证实了该理论,发现每条序列中至少含有1个开放阅读框,多者可达3~4个。理论上开放阅读框中是外显子区域,包含大量功能基因,但本研究筛选出的共13个特异性遗传标记经BLAST比对后未发现与之相似性很高的功能基因组,相似性基本上都低于15%,这一结果与利用RAPD和AFLP技术筛选中国对虾生长性状和抗病标记研究中的结论类似(刘萍等,200;孟宪红等,2005;何玉英等,2007;Yue et al,2005)。未发现相似性高的功能基因可能有以下几方面原因:首先,因为抗高氨氮和高pH功能由数量性状遗传位点控制,即由许多小的有效基因进行控制,而目前关于氨氮和pH的研究主要集中在毒性和免疫等影响方面,对于抗逆性相关基因研究报道很少;其次是目前对中国对虾基因组的功能基因尚未完全清楚,研究中获得的SRAP标记所对应功能基因可能未被认识。对于已找到的特异性分子标记进行BLAST分析时虽未发现与其同源性较高的功能基因,但可推测其与高氨氮或高pH耐受性密切相关。由于抗逆机理的复杂性,抗逆反应中的许多细节尚不清楚,亟须更深入的研究和探索;最后,所获得序列片段长度在600 bp内,而在NCBI所比对的已知基因序列长度为上万甚至十万级,所以会出现相似性低的现象。实验室正在对获得的SRAP序列片段进行大规模群体相关性验证研究,确定合适稳定的序列特征性片段扩增区域遗传标记,同时尽快利用共显性分子标记技术建立高密度、兼容性好的遗传连锁图谱,将获得的性状相关标记定位于连锁图谱,为分子标记辅助育种奠定坚实基础。
与传统育种方法相比,分子标记辅助育种具有缩短育种周期、提高育种效率以及受环境影响小等明显优势。本研究获得的SRAP分子标记可为选育适应力更强、适应范围更广的中国对虾新品系提供技术支持和理论参考,对于加速中国对虾的分子标记辅助育种进程具有基础推动作用。植物在逆境(干旱、盐碱、低温、病害胁迫下的生理变化和抗逆反应研究已基本清楚(齐宏飞等,2008),且随着分子生物学的发展,人们已从基因组成、表达调控和转录表达等方面进行了抗逆机理的研究,已经克隆出一些抗逆相关基因(周宜君等,2006)。而这些都是中国对虾乃至动物关于抗逆研究的方向和重点。
免责声明:以上内容源自网络,版权归原作者所有,如有侵犯您的原创版权请告知,我们将尽快删除相关内容。
















